Capítulo 2 Operações Básicas no R
Antes de começar a desenvolver o seu código, é necessário entender a forma de trabalhar com o R e o RStudio. Isso inclui os componentes da linguagem, os diferentes tipos de objetos e as operações que podem ser realizadas com base nos comandos existentes.
Neste capítulo iremos percorrer os passos iniciais sobre o ponto de vista de alguém que nunca trabalhou com o R e, possivelmente, nunca teve contato com outra linguagem de programação. Aqueles já familiarizados com o programa irão encontrar pouca informação nova e, portanto, sugiro a leitura da próxima seção. É recomendado, porém, que no mínimo esse usuário verifique os temas tratados para confirmar ou não o seu conhecimento sobre as funcionalidades do programa. Muitas das sugestões apresentadas aqui tem potencial de aumentar significativamente a sua produtividade no RStudio.
2.1 Como o R Funciona?
A maior dificuldade que um usuário iniciante possui ao começar a desenvolver rotinas com o R é a forma de trabalho. A nossa interação com computadores foi simplificada ao longo dos anos e atualmente estamos confortáveis com o formato de interação do tipo aponte e clique. Isto é, caso se queira efetuar alguma operação no computador, basta apontar o mouse para o local específico na tela e clicar um botão que realize tal operação. Uma série de passos nesse sentido permite a execução de tarefas complexas no computador. Mas não se engane, essa forma de interação no formato aponte e clique é apenas uma camada por cima do que realmente acontece no computador. Por trás de todo clique existe um comando sendo executado, seja na abertura de um arquivo pdf, direcionamento do browser para uma página na internet ou qualquer outra operação cotidiana de trabalho.
Enquanto esse formato de interação visual e motora tem seus benefícios ao facilitar e popularizar o uso de computadores, é pouco flexível e eficaz quando se trabalha com procedimentos computacionais. Ao conhecer os possíveis comandos disponíveis ao usuário, é possível criar um arquivo contendo alguns comandos em sequência e, futuramente, simplesmente pedir que o computador execute esse arquivo com os nossos procedimentos. Uma rotina de computador é nada mais do que um texto que instrui, de forma clara e sequencial, o que o computador deve fazer. Investe-se certo tempo para a criação do programa, porém, no futuro, esse irá executar sempre da mesma maneira o procedimento gravado. No médio e longo prazo, existe um ganho significativo de tempo entre o uso de uma rotina do computador e uma interface do tipo aponte e clique.
Além disso, o risco de erro humano na execução do procedimento é quase nulo, pois os comandos e a sua sequência estão registrados no arquivo texto e irão ser executados sempre da mesma maneira. Da mesma forma, esse aglomerado de comandos pode ser compartilhado com outras pessoas, as quais podem replicar os resultados em seus computadores. Essa é uma das grandes razões que justificam a popularização de programação na realização de pesquisa em dados. Todos os procedimentos executados podem ser replicados pelo uso de um script.
No uso do R, o ideal é mesclar o uso do mouse com a utilização de comandos. O R e o RStudio possuem algumas funcionalidades através do mouse, porém a sua capacidade é otimizada quando os utilizamos via inserção de comandos específicos. Quando um grupo de comandos é realizado de uma maneira inteligente, temos um script do R que deve preferencialmente produzir algo importante para nós no final de sua execução. Em Finanças e Economia, isso pode ser o valor atualizado de um portfólio de investimento, o cálculo de um índice de atividade econômica, a performance histórica de uma estratégia de investimento, o resultado de uma pesquisa acadêmica, entre diversas outras possibilidades.
O R também possibilita a exportação de arquivos, tal como figuras a serem inseridas em um relatório técnico ou informações em um arquivo texto. De fato, o próprio relatório técnico pode ser dinamicamente criado dentro do R através da tecnologia RMarkdown. Por exemplo, este livro que estás lendo foi escrito utilizando o pacote bookdown (Xie 2022), o qual é baseado em RMarkdown. O conteúdo do livro é compilado com a execução dos códigos e as suas saídas são registradas em texto. Todas as figuras e os dados do livro podem ser atualizados com a execução de um simples comando.
O produto final de trabalhar com R e RStudio será um script que produz elementos para um relatório de dados. Um bom exemplo de um código simples e polido pode ser encontrado neste link12. Abra-o e você verá o conteúdo de um arquivo com extensão .R que fará o download dos preços das ações de duas empresas e criará um gráfico e uma tabela. Ao terminar de ler o livro, você irá entender o que está acontecendo no código e como ele realiza o trabalho. Melhor ainda, você poderá melhorá-lo com novas funcionalidades e novas saídas. Caso esteja curioso em ver o script rodar, faça o seguinte: 1) instale R e RStudio no computador, 2) copie o conteúdo de texto do link para um novo script (“File” -> “New File” -> “R Script”), 3) salve-o com um nome qualquer e, finalizando, 4) pressione control + shift + enter para executar o script inteiro.
2.2 Objetos e Funções
No R, tudo é um objeto, e cada tipo de objeto tem suas propriedades. Por exemplo, o valor de um índice de inflação ao longo do tempo – em vários meses e anos – pode ser representado como um objeto do tipo vetor numérico. As datas em si, no formato YYYY-MM-DD (ano-mês-dia), podem ser representadas como texto (character) ou a própria classe Date. Por fim, podemos representar conjuntamente os dados de inflação e as datas armazenando-os em um objeto único do tipo dataframe, o qual nada mais é do que uma tabela com linhas e colunas. Todos esses objetos fazem parte do ecossistema do R e é através da manipulação destes que tiramos o máximo proveito do software.
Enquanto representamos informações do mundo real como diferentes classes no R, um tipo especial de objeto é a função, a qual representa um procedimento preestabelecido que está disponível para o usuário. O R possui uma grande quantidade de funções, as quais possibilitam que o usuário realize uma vasta gama de procedimentos. Por exemplo, os comandos básicos do R, não incluindo demais pacotes, somam um total de 1258 funções. Com base neles e outros iremos importar dados, calcular médias, testar hipóteses, limpar dados, e muito mais.
Cada função possui um próprio nome. Por exemplo, a função sort() é um procedimento que ordena valores valores utilizados como input. Caso quiséssemos ordnear os valores 2, 1, 3, 0, basta inserir no prompt o seguinte comando e apertar enter:
R> [1] 3 2 1 0O comando c(2, 1, 3, 0) combina os valores em um vetor (maiores detalhes sobre comando c serão dados em seção futura). Observe que a função sort é utilizada com parênteses de início e fim. Esses parênteses servem para destacar as entradas (inputs), isto é, as informações enviadas para a função produzir alguma coisa. Observe que cada entrada (ou opção) da função é separada por uma vírgula, tal como em MinhaFuncao(entrada01, entrada02, entrada03, ...). No caso do código anterior, note que usamos a opção decreasing = TRUE. Essa é uma instrução específica para a função sort ordenar de forma decrescente os elementos do vetor de entrada. Veja a diferença:
R> [1] 0 1 2 3O uso de funções está no coração do R e iremos dedicar grande parte do livro a elas. Por enquanto, essa breve introdução já serve o seu propósito. O principal é entender que uma função usa suas entradas para produzir algo de volta. Nos próximos capítulos iremos utilizar funções já existentes para as mais diferentes finalidades: baixar dados da internet, ler arquivos, realizar testes estatísticos e muito mais. No capítulo 8 iremos tratar deste assunto com maior profundidade, incluindo a forma de escrevermos nossas próprias funções.
2.3 O Formato Brasileiro
Antes de começar a explicar o uso do R e RStudio, é importante ressaltar algumas regras de formatação de números e códigos para o caso brasileiro.
Decimal: O decimal no R é definido pelo ponto (.), tal como em 2.5 e não vírgula, como em 2,5. Esse é o padrão internacional, e a diferença para a notação brasileira gera muita confusão. Alguns softwares, por exemplo o Microsoft Excel, fazem essa conversão automaticamente no momento da importação dos dados. Porém isso não ocorre na maioria dos casos. Como regra geral, utilize vírgulas apenas para separar os termos de entradas em uma função (veja exemplo de seção anterior com função sort). Em nenhuma situação deve-se utilizar a vírgula como separador de casas decimais. Mesmo quando estiver exportando dados, sempre dê prioridade para o formato internacional, pois esse será compatível com a grande maioria dos dados e facilitará o uso do software.
Caracteres latinos: Devido ao seu padrão internacional, o R apresenta problemas para entender caracteres latinos, tal como cedilha e acentos. Caso possa evitar, não utilize esses tipos de caracteres no código para nomeação de variáveis ou arquivos. Nos objetos de classe texto (character), é possível utilizá-los desde que a codificação do objeto esteja correta (UTF-8 ou Latin1). Assim, recomenda-se que o código do R seja escrito na língua inglesa. Isso automaticamente elimina o uso de caracteres latinos e facilita a usabilidade do código por outras pessoas que não entendam a língua portuguesa. Destaca-se que essa foi a escolha utilizada para o livro. Os nomes dos objetos nos exemplos estão em inglês, assim como também todos os comentários do código.
Formato das datas: Datas no R são formatadas de acordo com a norma ISO 8601, seguindo o padrão YYYY-MM-DD, onde YYYY é o ano em quatro números, MM é o mês e DD é o dia. Por exemplo, uma data em ISO 8601 é 2022-11-23. No Brasil, as datas são formatadas como DD/MM/YYYY. Reforçando a regra, sempre dê preferência ao padrão internacional. Vale salientar que a conversão entre um formato e outro é bastante fácil e será apresentada em capítulo futuro.
No momento de instalação do R, diversas informações sobre o formato local do seu computador são importadas do seu sistema operacional. Para saber qual o formato que o R está configurado localmente, digite o seguinte comando no prompt (canto esquerdo inferior do RStudio) e aperte enter:
# get local format
Sys.localeconv()R> decimal_point thousands_sep grouping
R> "." "" ""
R> int_curr_symbol currency_symbol mon_decimal_point
R> "BRL " "R$" ","
R> mon_thousands_sep mon_grouping positive_sign
R> "." "\003\003" ""
R> negative_sign int_frac_digits frac_digits
R> "-" "2" "2"
R> p_cs_precedes p_sep_by_space n_cs_precedes
R> "1" "1" "1"
R> n_sep_by_space p_sign_posn n_sign_posn
R> "1" "1" "1"A saída de Sys.localeconv() mostra como o R interpreta pontos decimais e o separador de milhares, entre outras coisas. Como você pode ver no resultado anterior, este livro foi compilado usando a notação brasileira de moeda (BRL/R$), mas usa a formatação internacional – o ponto (.) – para decimais.
Muito cuidado ao modificar o formato que o R interpreta os diferentes símbolos e notações. Como regra de bolso, caso precisar usar algum formato específico, faça-o isoladamente dentro do contexto do código. Evite mudanças permanentes pois nunca se sabe onde tais formatos estão sendo usados. Evite, assim, surpresas desagradáveis no futuro.
2.4 Tipos de Arquivos
Assim como outros programas, o R possui um ecossistema de arquivos e cada extensão tem uma finalidade diferente. A seguir apresenta-se uma descrição de diversas extensões de arquivos. Os itens da lista estão ordenados por ordem de importância e uso. Note que omitimos arquivos de figuras tal como .png, .jpg, .gif entre outros, pois estes não são exclusivos do R.
Arquivos com extensão .R: Representam arquivos do tipo texto contendo diversas instruções para o R. Esses são os arquivos que conterão o código da pesquisa e onde passaremos a maior parte do tempo. Também pode ser chamado de um script ou rotina de pesquisa. Como sugestão, pode-se dividir toda uma pesquisa em etapas e, para cada, nomear script correspondente. Exemplos: 01-Get-Data.R, 02-Clean-data.R, 03_Estimate_Models.R.
Arquivos com extensão .RData e .rds: armazenam dados nativos do R. Esses arquivos servem para salvar objetos do R em um arquivo no disco rígido do computador para, em sessão futura, serem novamente carregados. Por exemplo, podes guardar o resultado de uma pesquisa em uma tabela, a qual é salva em um arquivo com extensão .RData ou .rds. Exemplos: Raw-Data.RData, Table-Results.rds.
Arquivos com extensão .Rmd, .md e .Rnw: São arquivos relacionados a tecnologia Rmarkdown. O uso desses arquivos permite a criação de documentos onde texto e código são integrados.
Arquivos com extensão .Rproj: Contém informações para a edição de projetos no RStudio. O sistema de projetos do RStudio permite a configuração customizada do projeto e também facilita a utilização de ferramentas de controle de código, tal como controle de versões. O seu uso, porém, não é essencial. Para aqueles com interesse em conhecer esta funcionalidade, sugiro a leitura do manual do RStudio13. Uma maneira simples de entender os tipos de projetos disponíveis é, no RStudio, clicar em “File”, “New project”, “New Folder” e assim deve aparecer uma tela com todos os tipos possíveis de projetos no RStudio. Exemplo: My-Dissertation-Project.Rproj.
2.5 Explicando a Tela do RStudio
Após instalar os dois programas, R e RStudio, Procure o ícone do RStudio na área de trabalho ou via menu Iniciar. Note que a instalação do R inclui um programa de interface e isso muitas vezes gera confusão. Verifique que estás utilizado o software correto. A janela resultante deve ser igual a figura 2.1, apresentada a seguir.
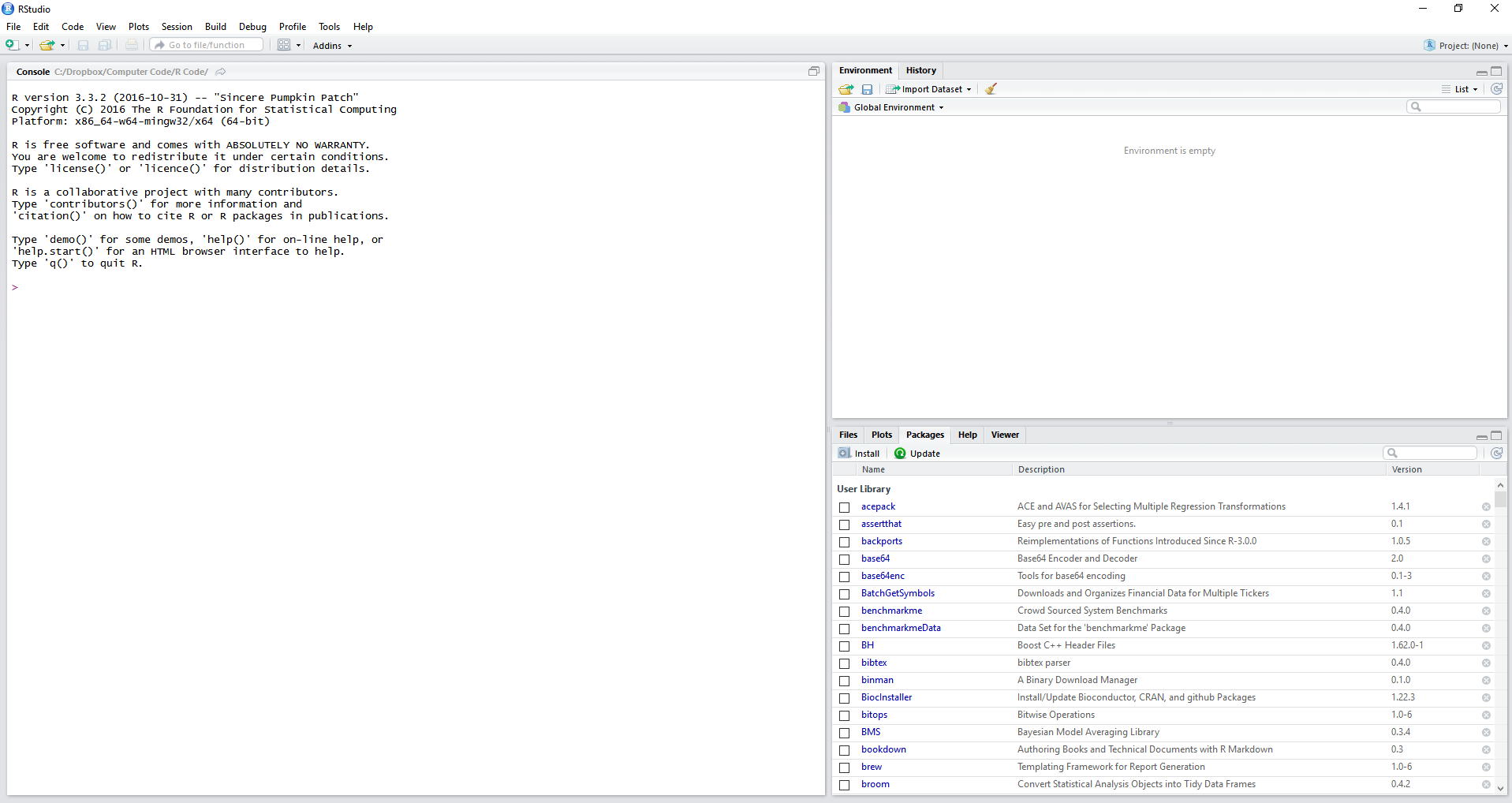
Figura 2.1: A tela do RStudio
Observe que o RStudio automaticamente detectou a instalação do R e inicializou a sua tela no lado esquerdo. Caso não visualizar uma tela parecida ou chegar em uma mensagem de erro indicando que o R não foi encontrado, repita os passos de instalação do capítulo anterior (seção 1.4).
Como um primeiro exercício, clique em File, New File e R Script. Após, um editor de texto deve aparecer no lado esquerdo da tela do RStudio. É nesse editor que iremos inserir os nossos comandos, os quais são executados de cima para baixo, na mesma direção em que normalmente o lemos. Note que essa direção de execução introduz uma dinâmica de recursividade: cada comando depende do comando executado nas linhas anteriores. Após realizar os passos definidos anteriormente, a tela resultante deve ser semelhante à apresentada na figura 2.2.
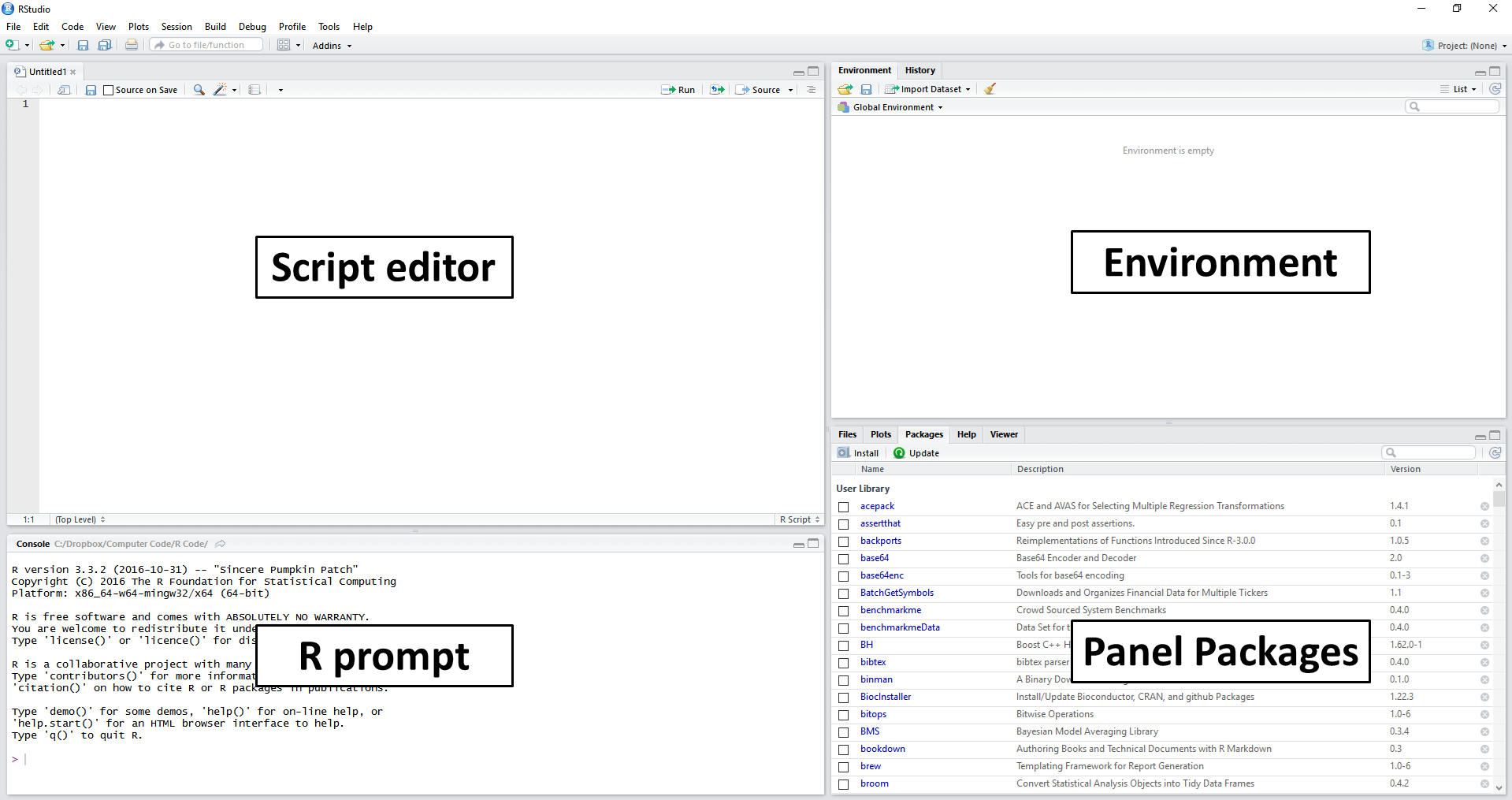
Figura 2.2: Explicando a tela do RStudio
Uma sugestão importante aqui é modificar o esquema de cores do RStudio para uma configuração de tela escura. Não é somente uma questão estética mas sim de prevenção e melhoria de sua saúde física. Possivelmente irás passar demasiado tempo na frente do computador. Assim, vale a pena modificar as cores da interface para aliviar seus olhos do constante brilho da tela. Dessa forma, conseguirás trabalhar por mais tempo, sem forçar a sua visão. Podes configurar o esquema de cores do RStudio indo na opção Tools, Global Options e então em Appearance. Um esquema de cores escuras que pessoalmente gosto e sugiro é o Ambience.
Após os passos anteriores, a tela do RStudio deve estar semelhante a Figura 2.2, com os seguintes itens/painéis:
Editor de scripts (Script editor): localizado no lado esquerdo e acima da tela. Esse painel é utilizado para escrever código e é onde passaremos a maior parte do tempo.
Console do R (R prompt): localizado no lado esquerdo e abaixo do editor de scripts. Apresenta o prompt do R, o qual também pode ser utilizado para executar comandos. A principal função do prompt é testar código e apresentar os resultados dos comandos inseridos no editor de scripts.
Área de trabalho (Environment): localizado no lado direito e superior da tela. Mostra todos os objetos, incluindo variáveis e funções atualmente disponíveis para o usuário. Observe também a presença do painel History, o qual mostra o histórico dos comandos já executados.
Pacotes (Panel Packages): mostra os pacotes instalados e carregados pelo R. Um pacote é nada mais que um módulo no R, cada qual com sua finalidade específica. Observe a presença de quatro abas: Files, para carregar e visualizar arquivos do sistema; Plots, para visualizar figuras; Help, para acessar o sistema de ajuda do R e Viewer, para mostrar resultados dinâmicos e interativos, tal como uma página da internet.
Como um exercício introdutório, vamos inicializar duas variáveis. Dentro do console do R (lado esquerdo inferior), digite os seguintes comandos e aperte enter ao final de cada linha. O símbolo <- é nada mais que a junção de < com -. O símbolo ' representa uma aspa simples e sua localização no teclado Brasileiro é no botão abaixo do escape (esc), lado esquerdo superior do teclado.
# set x and y
x <- 1
y <- 'my text'Após a execução, dois objetos devem aparecer no painel Environment, um chamado x com o valor 1, e outro chamado y com o conjunto de caracteres 'my text'. O histórico de comandos na aba History também foi atualizado com os comandos utilizados anteriormente.
Agora, vamos mostrar na tela os valores de x. Para isso, digite o seguinte comando no prompt e aperte enter novamente:
# print x
print(x)R> [1] 1A função print é uma das principais funções para mostrarmos valores no prompt do R. O texto apresentado como [1] indica o índice do primeiro número da linha. Para verificar isso, digite o seguinte comando, o qual irá mostrar vários números na tela:
# print vector from 50 to 100
print(50:100)R> [1] 50 51 52 53 54 55 56 57 58 59 60 61 62 63
R> [15] 64 65 66 67 68 69 70 71 72 73 74 75 76 77
R> [29] 78 79 80 81 82 83 84 85 86 87 88 89 90 91
R> [43] 92 93 94 95 96 97 98 99 100Nesse caso, utilizamos o símbolo : em 50:100 para criar uma sequência iniciando em 50 e terminando em 100. Observe que temos valores encapsulados por colchetes ([]) no lado esquerda da tela. Esses representam os índices do primeiro elemento apresentado na linha. Por exemplo, o décimo quinto elemento do vetor criado é o valor 64.
2.6 Pacotes do R
Um dos grandes benefícios do uso do R é o seu acervo de pacotes. Esses representam um conjunto de procedimentos agrupados em uma coleção de funções e voltados para a resolução de um problema qualquer. O R tem em sua essência uma filosofia de colaboração. Usuários disponibilizam os seus códigos para outras pessoas utilizarem. E, mais importante, todos os pacotes são gratuitos, assim como o R. Por exemplo, considere um caso em que está interessado em baixar dados da internet sobre o desemprego histórico no Brasil. Para isso, basta procurar e instalar o pacote específico que realiza esse procedimento.
Esses pacotes podem ser instalados de diferentes fontes, com as principais sendo CRAN (The Comprehensive R Archive Network) e Github. A cada dia aumenta a quantidade e diversidade de pacotes existentes para o R. O próprio autor deste livro possui diversos pacotes disponíveis no CRAN, cada um para resolver algum problema diferente. Na grande maioria, são pacotes para importar e organizar dados financeiros.
O CRAN é o repositório oficial do R e é livre. Qualquer pessoa pode enviar um pacote e todo código enviado está disponível na internet. Existe, porém, um processo de avaliação que o código passa e certas normas rígidas devem ser respeitadas sobre o formato do código, o manual do usuário e a forma de atualização do pacote. Para quem tiver interesse, um tutorial claro e fácil de seguir é apresentado no site http://r-pkgs.had.co.nz/intro.html. As regras completas estão disponíveis no site do CRAN - https://cran.r-project.org/web/packages/policies.html. A adequação do código a essas normas é responsabilidade do desenvolvedor e gera um trabalho significativo, principalmente na primeira submissão.
A lista completa de pacotes disponíveis no CRAN, juntamente com uma breve descrição, pode ser acessada no link packages do site do R - https://cran.r-project.org/. Uma maneira prática de verificar a existência de um pacote para um procedimento específico é carregar a página anterior e procurar no seu navegador de internet a palavra-chave que define o seu procedimento. Caso existir o pacote com a palavra-chave, a procura acusará o encontro do termo na descrição do pacote.
Outra fonte importante para o encontro de pacotes é o Task Views, em que são destacados os principais pacotes de acordo com a área e o tipo de uso. Veja a tela do Task Views na Figura \(\ref{fig:TaskViews}\).

Figura 2.3: Tela do Task Views
Ao contrário do CRAN, o Github não possui restrição quanto ao código enviado e, devido a isso, tende a ser escolhido como ambiente de compartilhamento de código. A responsabilidade de uso, porém, é do próprio usuário. Na prática, é muito comum os desenvolvedores manterem uma versão em desenvolvimento no Github e outra oficial no CRAN. Quando a versão em desenvolvimento atinge um estágio de maturidade, a mesma é enviada ao CRAN.
O mais interessante no uso de pacotes é que estes podem ser acessados e instalados diretamente no R via a internet. Para saber qual é a quantidade atual de pacotes no CRAN, digite e execute os seguintes comandos no prompt:
# find current available packages
df_cran_pkgs <- available.packages()
# get size of matrix
n_cran_pkgs <- nrow(df_cran_pkgs)
# print it
print(n_cran_pkgs)R> [1] 18877Atualmente, 2022-11-23 10:51:29, existem 18877 pacotes disponíveis nos servidores do CRAN.
Também se pode verificar a quantidade de pacotes localmente instalados com o comando installed.packages():
# get number of local (installed) packages
n_local_pkgs <- nrow(installed.packages())
# print it
print(n_local_pkgs)R> [1] 531Nesse caso, o computador em que o livro foi escrito possui 531 pacotes do R instalados. Note que, apesar do autor ser um experiente programador do R, apenas uma pequena fração do pacotes disponíveis no CRAN está sendo usada! A diversidade dos pacotes é gigantesca.
2.6.1 Instalando Pacotes do CRAN
Para instalar um pacote, basta utilizar o comando install.packages. Como exemplo, vamos instalar um pacote que será utilizado nos capítulos futuros, o readr:
# install pkg readr
install.packages('readr')Copie e cole este comando no prompt e pronto! O R irá baixar os arquivos necessários e instalar o pacote readr e suas dependências. Após isto, as funções relativas ao pacote estarão prontas para serem usadas após o carregamento do módulo (detalhes a seguir). Observe que definimos o nome do pacote na instalação como se fosse texto, com o uso das aspas ("). Caso o pacote instalado seja dependente de outros pacotes, o R automaticamente instala todos módulos faltantes. Assim, todos os requerimentos para o uso do respectivo pacote já serão satisfeitos e tudo funcionará perfeitamente. É possível, porém, que um pacote tenha uma dependência externa. Como um exemplo, o pacote RndTexExams depende da existência de uma instalação do LaTex. Geralmente essa é anunciada na sua descrição e um erro é sinalizado na execução do programa quando o LaTex não é encontrado. Fique atento, portanto, a esses casos.
Aproveitando o tópico, sugiro que o leitor já instale todos os pacotes do tidyverse com o seguinte código:
# install pkgs from tidyverse
install.packages('tidyverse')O tidyverse é um conjunto de pacotes voltados a data science e com uma sintaxe própria e consistente, voltada a praticabilidade. Verás que, em uma instalação nova do R, o tidyverse depende de uma grande quantidade de pacotes.
2.6.2 Instalando Pacotes do Github
Para instalar um pacote diretamente do Github, é necessário instalar antes o pacote devtools, disponível no CRAN:
# install devtools
install.packages('devtools')Após isto, utilize função devtools::install_github para instalar um pacote diretamente do Github. Note que o símbolo :: indica que função install_github pertence ao pacote devtools. Com esta particular sintaxe, não precisamos carregar todo o pacote para utilizar apenas uma função.
No exemplo a seguir instalamos a versão em desenvolvimento do pacote ggplot2, cuja versão oficial também está disponível no CRAN:
# install ggplot2 from github
devtools::install_github("hadley/ggplot2")Observe que o nome do usuário do repositório também é incluído. No caso anterior, o nome hadley pertence ao desenvolvedor do ggplot2, Hadley Wickham. No decorrer do livro notará que esse nome aparecerá diversas vezes, dado que Hadley é um prolífico e competente desenvolvedor de diversos pacotes do R e do tidyverse.
Um aviso aqui é importante. Os pacotes do github não são moderados. Qualquer pessoa pode enviar código para lá e o conteúdo não é checado de forma independente. Nunca instale pacotes do github sem conhecer os autores. Apesar de improvável – nunca aconteceu comigo por exemplo – é possível que esses possuam algum código malicioso.
2.6.3 Carregando Pacotes
Dentro de uma rotina de pesquisa, utilizamos a função library para carregar um pacote na nossa sessão do R. Ao fecharmos o RStudio ou então iniciar uma nova sessão do R, os pacotes são descarregados. Vale salientar que alguns pacotes, tal como o base e o stats, são inicializados automaticamente a cada nova sessão. A grande maioria, porém, deve ser carregada no início dos scripts. Veja o exemplo a seguir:
A partir disso, todas as funções do pacote estarão disponíveis para o usuário. Note que não é necessário utilizar aspas (") ao carregar o pacote. Caso utilize uma função específica do pacote e não deseje carregar todo ele, pode fazê-lo através do uso do símbolo especial ::, conforme o exemplo a seguir.
# call fct fortune() from pkg fortune
fortunes::fortune(10)R>
R> Overall, SAS is about 11 years behind R and S-Plus in
R> statistical capabilities (last year it was about 10 years
R> behind) in my estimation.
R> -- Frank Harrell (SAS User, 1969-1991)
R> R-help (September 2003)Nesse caso, utilizamos a função fortune do próprio pacote fortunes, o qual mostra na tela uma frase possivelmente engraçada escolhida do mailing list do R. Nesse caso, selecionamos a mensagem número 10. Se não tiver disponível o pacote, o R mostrará a seguinte mensagem de erro:
R> Error in library("fortune") : there is no package called "fortune"Para resolver, utilize o comando install.packages("fortunes") para instalar o pacote no seu computador. Execute o código fortunes::fortune(10) no prompt para confirmar a instalação. Toda vez que se deparar com essa mensagem de erro, deves instalar o pacote que está faltando.
Outra maneira de carregar um pacote é através da função require. Essa tem um comportamento diferente da função library e deve ser utilizada dentro da definição de funções ou no teste do carregamento do pacote. Caso o usuário crie uma função customizada que necessite de procedimentos de um pacote em particular, o mesmo deve carregar o pacote no escopo da função. Por exemplo, veja o código a seguir, em que criamos uma função dependente do pacote quantmod:
my_fct <- function(x){
require(quantmod)
df <- getSymbols(x, auto.assign = F)
return(df)
}Nesse caso, a função getSymbols faz parte do pacote quantmod. Não se preocupe agora com a estrutura utilizada para criar uma função no R. Essa será explicada em capítulo futuro.
Uma precaucão que deve sempre ser tomada quando se carrega um pacote
é um possível conflito de funções. Por exemplo, existe
uma função chamada filter no pacote dplyr e
também no pacote stats. Caso carregarmos ambos pacotes e
chamarmos a função filter no escopo do código, qual delas o
R irá usar? Pois bem, a preferência é sempre para o último
pacote carregado. Esse é um tipo de problema que pode gerar
muita confusão. Felizmente, note que o próprio R acusa um conflito de
nome de funções no carregamento do pacote. Para testar, inicie uma nova
sessão do R e carregue o pacote dplyr. Verás que uma
mensagem indica haver dois conflitos com o pacote stats e
quatro com pacote o base.
2.6.4 Atualizando Pacotes
Ao longo do tempo, é natural que os pacotes disponibilizados no CRAN sejam atualizados para acomodar novas funcionalidades ou se adaptar a mudanças em suas dependências. Assim, é recomendável que os usuários atualizem os seus pacotes instalados para uma nova versão através da internet. Esse procedimento é bastante fácil. Uma maneira direta de atualizar pacotes é clicar no botão update no painel de pacotes no canto direito inferior do RStudio, conforme mostrado na figura 2.4.
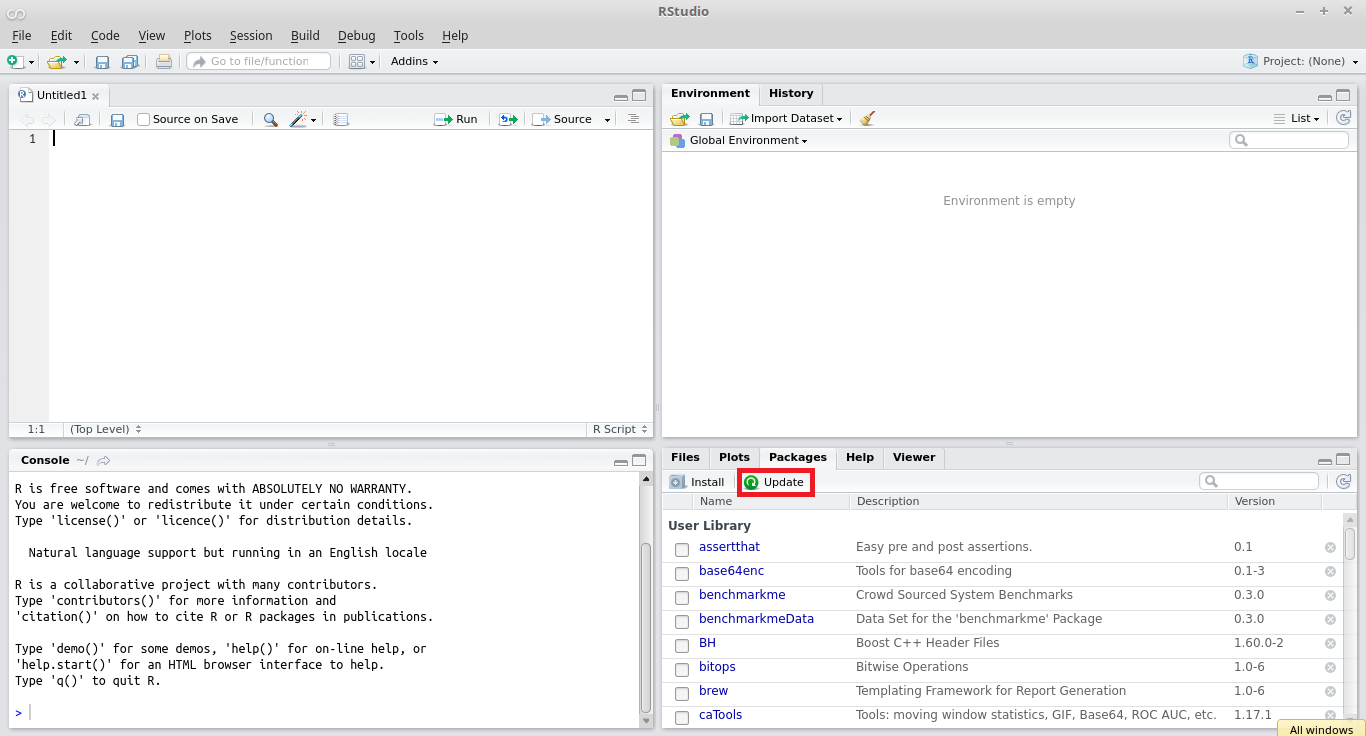
Figura 2.4: Atualizando pacotes no R
A atualização de pacotes através do prompt também é possível. Para isso, basta utilizar o comando update.packages, conforme mostrado a seguir.
O comando update.packages() compara a versão dos pacotes instalados em relação a versão disponível no CRAN. Caso tiver alguma diferença, a nova versão é instalada. Após a execução do comando, todos os pacotes estarão atualizados com a versão disponível nos servidores do CRAN.
Versionamento de pacotes é extremamente importante para manter a reproducibilidade do código. Apesar de ser raro de acontecer, é possível que a atualização de um pacote no R modifique, para os mesmos dados, resultados já obtidos anteriormente. Tenho uma experiência particularmente memorável quando um artigo científico retornou da revisão e, devido a atualização de um dos pacotes, não consegui reproduzir os resultados apresentados no artigo. No final deu tudo certo, mas o trauma fica.
Uma solução para este problema é congelar as versões dos pacotes para
cada projeto usando a ferramenta packrat do RStudio. Em
resumo, o packrat faz cópias locais dos pacotes utilizados
no projeto, os quais têm preferência aos pacotes do sistema. Assim, se
um pacote for atualizado no sistema, mas não no projeto, o código R vai
continuar usando a versão mais antiga e seu código sempre rodará nas
mesmas condições.
2.7 Executando Códigos em um Script
Agora, vamos juntar todos os códigos digitados anteriormente e colar na tela do editor (lado esquerdo superior), assim como mostrado a seguir:
Após colar todos os comandos no editor, salve o arquivo .R em alguma pasta pessoal. Esse arquivo, o qual no momento não faz nada de especial, registrou os passos de um algoritmo simples que cria dois objetos e mostra os seus valores. Futuramente esse irá ter mais forma, com a importação de dados, manipulação e modelagem dos mesmos e saída de tabelas e figuras.
No RStudio existem alguns atalhos predefinidos para executar códigos que economizam bastante tempo. Para executar um script inteiro, basta apertar control + shift + s. Esse é o comando source. Com o RStudio aberto, sugiro testar essa combinação de teclas e verificar como o código digitado anteriormente é executado, mostrando os valores no prompt do R. Visualmente, o resultado deve ser próximo ao apresentado na figura 2.5.
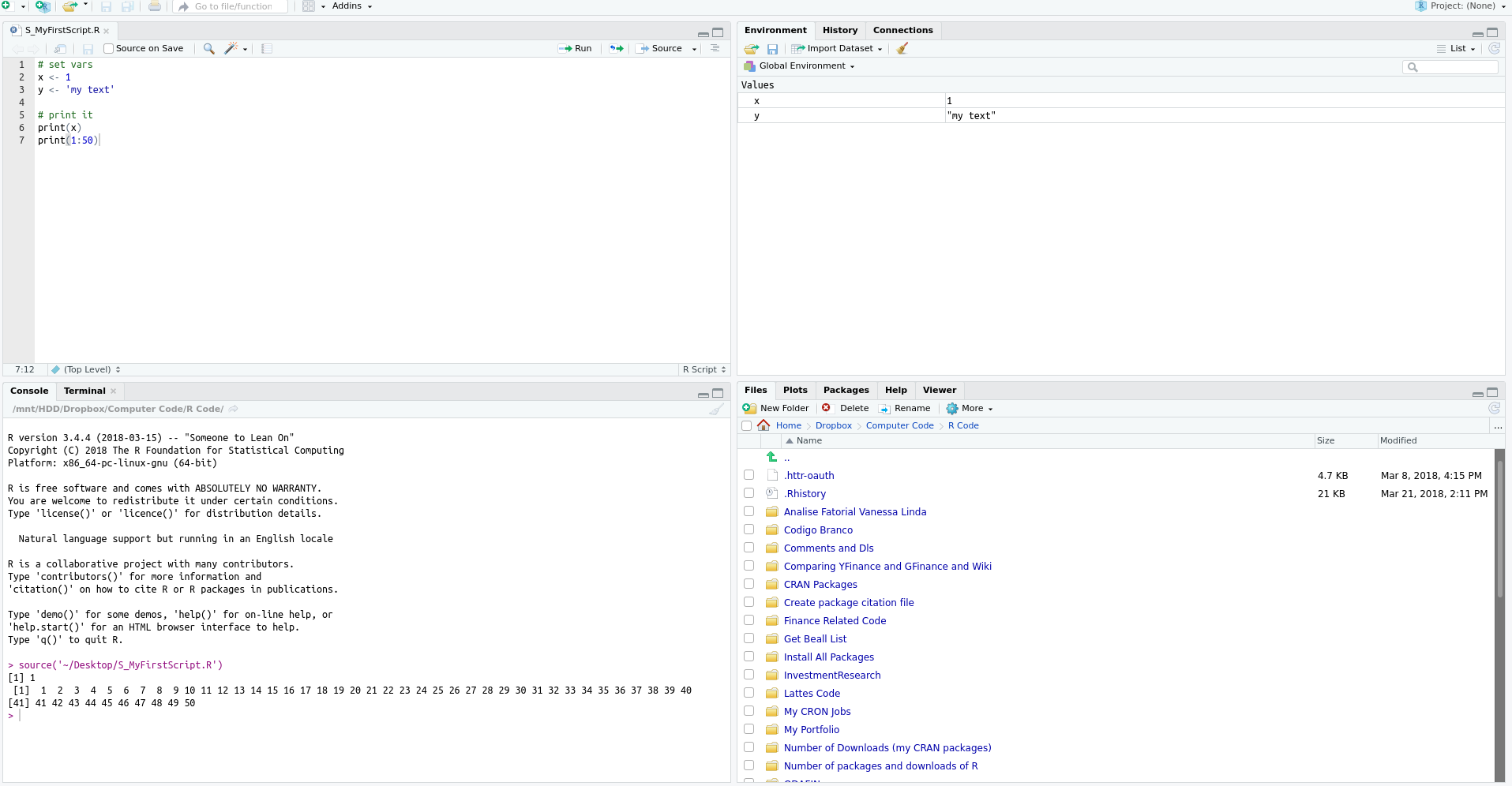
Figura 2.5: Exemplo de Rotina no R
Outro comando muito útil é a execução por linha. Nesse caso não é executado todo o arquivo, mas somente a linha em que o cursor do mouse se encontra. Para isto, basta apertar control+enter. Esse atalho é bastante útil no desenvolvimento de rotinas pois permite que cada linha seja testada antes de executar o programa inteiro. Como um exemplo de uso, aponte o cursor para a linha print(x) e pressione control + enter. Verás que o valor de x é mostrado na tela do prompt. A seguir destaco esses e outros atalhos do RStudio, os quais também são muito úteis.
- control+shift+s executa o arquivo atual do RStudio, sem mostrar comandos no prompt (sem eco – somente saída);
- control+shift+enter: executa o arquivo atual, mostrando comandos na tela (com eco – código e saída);
- control+enter: executa a linha selecionada, mostrando comandos na tela;
- control+shift+b: executa os códigos do início do arquivo até a linha atual onde o cursor se encontra;
- control+shift+e: executa os códigos da linha onde o cursor se encontra até o final do arquivo.
Sugere-se que esses atalhos sejam memorizados e utilizados. Isso facilita bastante o uso do programa. Para aqueles que gostam de utilizar o mouse, uma maneira alternativa para rodar o código do script é apertar o botão source, localizado no canto direito superior do editor de rotinas. Isto é equivalente ao atalho control+shift+s.
Porém, no mundo real de programação, poucos são os casos em que uma análise de dados é realizada por um script apenas. Como uma forma de organizar o código, pode-se dividir o trabalho em N scripts diferentes, onde um deles é o “mestre”, responsável por rodar os demais.
Neste caso, para executar os scripts em sequência, basta chamá-los no script mestre com o comando source, como no código a seguir:
# Import all data
source('01-import-data.R')
# Clean up
source('02-clean-data.R')
# Build tables
source('03-build-table.R')Nesse caso, o código anterior é equivalente a abrirmos e executarmos (control + shift + s) cada um dos scripts sequencialmente.
Como podemos ver, existem diversas maneiras de executar uma rotina de pesquisa. Na prática, porém, iras centralizar o uso em dois comandos apenas: control+shift+s para rodar o script inteiro e control+enter para rodar por linha.
2.8 Testando Código
O desenvolvimento de códigos em R segue um conjunto de etapas. Primeiro você escreverá uma nova linha de comando em uma rotina. Essa linha será testada com o atalho control + enter, verificando-se a ocorrência de erros e as saídas na tela. Caso não houver erro e o resultado for igual ao esperado, parte-se para a próxima linha de código.
Um ciclo de trabalho fica claro, a escrita do código da linha atual é seguida pela execução, seguido da verificação de resultados, modificação caso necessário e assim por diante. Esse é um processo normal e esperado. Dado que uma rotina é lida e executada de cima para baixo, você precisa ter certeza de que cada linha de código está corretamente especificada antes de passar para a próxima.
Quando você está tentando encontrar um erro em um script preexistente, o R oferece algumas ferramentas para controlar e avaliar sua execução. Isso é especialmente útil quando você possui um código longo e complicado. A ferramenta de teste mais simples e fácil de utilizar que o RStudio oferece é o ponto de interrupção do código. No RStudio, você pode clicar no lado esquerdo do editor e aparecerá um círculo vermelho, como na Figura 2.6.
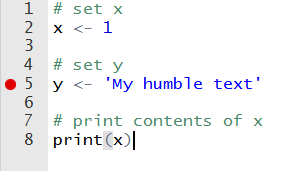
Figura 2.6: Exemplo de debug
O círculo vermelho indica um ponto de interrupção do código que forçará o R a pausar a execução nessa linha. Quando a execução atinge o ponto de interrupção, o prompt mudará para browser[1]> e você poderá verificar o conteúdo dos objetos. No console, você tem a opção de continuar a execução para o próximo ponto de interrupção ou interrompê-la. O mesmo resultado pode ser alcançado usando a função browser. Dê uma olhada:
O resultado prático do código anterior é o mesmo que utilizar o círculo vermelho do RStudio, figura 2.6. Porém, o uso do browser permite mais controle sobre onde a execução deve ser pausada. Como um teste, copie e cole o código anterior no RStudio, salve em um novo script e execute com Control + Shift + S. Para sair do ambiente de depuramento (debug), aperte enter no prompt do RStudio.
2.9 Criando Objetos Simples
Um dos comandos mais básicos no R é a definição de objetos. Como foi mostrado nas seções anteriores, pode-se definir um objeto com o uso do comando <-, o qual, para o português, é traduzido para o verbo defina (assign em inglês). Considere o seguinte código:
# set x
my_x <- 123
# set x, y and z in one line
my_x <- 1 ; my_y <- 2; my_z <- 3Lê-se esse código como x é definido como 123. A direção da seta define onde o valor será armazenado. Por exemplo, utilizar 123 -> my_x também funcionaria, apesar de ser uma sintaxe pouco utilizada ou recomendada. Note que também é possível escrever diversos comandos na mesma linha com o uso da semi-vírgula (;).
O uso do símbolo <- para a definição de objetos é
específico do R. Na época da concepção da linguagem S, de onde
o R foi baseado, existiam teclados com uma tecla específica que definia
diretamente o símbolo de seta. Teclados contemporâneos, porém, não
possuem mais esta configuração. Uma alternativa é utilizar o atalho para
o símbolo, o qual, no Windows, é definido por alt + -.
É possível também usar o símbolo = para definir objetos assim como o <-. Saliento que esta é prática comum em outras linguagens de programação. Porém, no ecosistema do R, a utilização do = com esse fim específico não é recomendada. O símbolo de igualdade tem o seu uso especial e resguardado na definição de argumentos de uma função tal como sort(x = 1:10, decreasing = TRUE).
O nome dos objetos é importante no R. Tirando alguns casos específicos, o usuário pode nomear os objetos como quiser. Essa liberdade, porém, pode ser um problema. É desejável sempre dar nomes curtos que façam sentido ao conteúdo do objeto e que sejam simples de entender. Isso facilita o entendimento do código por outros usuários e faz parte das normas sugeridas para a estruturação do código. Observe que todos os objetos criados nesse livro possuem nomenclatura em inglês e formatação específica, onde espaços entre substantivos e verbos são substituídos por traço baixo, tal como em my_x e my_csv_file. Aqui, o mais importante é a consistência do formato. Sempre mantenha o mesmo padrão em todo o código. No capítulo 13 vamos ir mais a fundo nesta questão de estrutura de código.
O R executa o código procurando objetos e funções disponíveis no seu ambiente de trabalho (enviromnent). Se tentarmos acessar um objeto que não existe, o R irá retornar uma mensagem de erro:
print(z)R> Error in print(z): object 'z' not foundIsso ocorre pois o objeto z não existe na sessão atual do R. Se criarmos uma variável z como z <- 123 e repetirmos o comando print(z), não teremos a mesma mensagem de erro.
Um ponto importante aqui é a definição de objetos de classes diferentes com o uso de símbolos específicos. O uso de aspas duplas (" ") ou simples (' ') define objetos da classe texto enquanto números são definidos pelo próprio valor. Conforme será mostrado, cada objeto no R tem uma classe e cada classe tem um comportamento diferente. Portanto, objetos criados com o uso de aspas pertencem à classe character. Podemos confirmar isso via código:
# set vars
x <- 1
y <- '1'
# display classes
class(x)R> [1] "numeric"
class(y)R> [1] "character"As saídas anteriores mostram que a variável x é do tipo numérico, enquanto a variável y é do tipo texto (character). Ambas fazem parte das classes básicas de objetos no R. Por enquanto, este é o mínimo que deves saber para avançar nos próximos capítulos. Iremos estudar este assunto mais profundamente no capítulo 7.
2.10 Criando Vetores
Nos exemplos anteriores criamos objetos simples tal como x <- 1 e x <- 'abc'. Enquanto isso é suficiente para demonstrar os comandos básicos do R, na prática tais comandos são bastante limitados, uma vez que um problema real de análise de dados certamente irá ter um maior volume de informações do mundo real.
Um dos procedimentos mais utilizados no R é a criação de vetores atômicos. Esses são objetos que guardam uma série de elementos. Todos os elementos de um vetor atômico devem possuir a mesma classe, o que justifica a sua propriedade atômica. Um exemplo seria representar no R uma série de preços diários de uma ação. Tal série possui vários valores numéricos que formam um vetor da classe numérica.
Vetores atômicos são criados no R através do uso do comando c, o qual é oriundo do verbo em inglês combine. Por exemplo, caso eu quisesse combinar os valores 1, 2 e 3 em um objeto/vetor, eu poderia fazê-lo através do seguinte comando:
R> [1] 1 2 3Esse comando funciona da mesma maneira para qualquer número de elementos. Caso necessário, poderíamos criar um vetor com mais elementos simplesmente adicionando valores após o 3, tal como em x <- c(1, 2, 3, 4, 5).
O uso do comando c não é exclusivo para vetores numéricos. Por exemplo, poderíamos criar um vetor de outra classe de dados, tal como character:
R> [1] "text 1" "text 2" "text 3" "text 4"A única restrição no uso do comando c é que todos os itens do vetor tenham a mesma classe. Se inserirmos dados de classes diferentes, o R irá tentar transformar os itens para a mesma classe seguindo uma lógica própria, onde a classe mais complexa sempre tem preferência. Caso ele não consiga transformar todos os elementos para uma classe só, uma mensagem de erro será retornada. Observe no próximo exemplo como os valores numéricos no primeiro e segundo elemento de x são transformados para a classe de caracteres.
R> [1] "numeric"R> [1] "character"Outra utilização do comando c é a combinação de vetores. De fato, isto é exatamente o que fizemos ao executar o código c(1, 2, 3). Neste caso, cada vetor possuía um elemento. Podemos realizar o mesmo com vetores maiores. Veja a seguir:
R> [1] 1 2 3 4 5Portanto, o comando c possui duas funções principais: criar e combinar vetores.
2.11 Conhecendo os Objetos Criados
Após a execução de diversos comandos no editor ou prompt, é desejável saber quais são os objetos criados pelo código. É possível descobrir essa informação simplesmente olhando para o lado direito superior do RStudio, na aba da área de trabalho. Porém, existe um comando que sinaliza a mesma informação no prompt. Com o fim de saber quais são as variáveis atualmente disponíveis na memória do R, pode-se utilizar o comando ls. Observe o exemplo a seguir:
# set vars
x <- 1
y <- 2
z <- 3
# show current objects
ls()R> [1] "x" "y" "z"Os objetos x, y e z foram criados e estavam disponíveis no ambiente de trabalho atual, juntamente com outros objetos. Para descobrir os valores dos mesmos, basta digitar os nomes dos objetos e apertar enter no prompt:
xR> [1] 1
yR> [1] 2
zR> [1] 3Destaca-se que digitar o nome do objeto na tela tem o mesmo resultado que utilizar a função print. De fato, ao executar o nome de uma variável, internamente o R passa esse objeto para a função print.
No R, conforme já mostrado, todos os objetos pertencem a alguma classe. Para descobrir a classe de um objeto, basta utilizar a função class. Observe no exemplo a seguir que x é um objeto da classe numérica e y é um objeto da classe de texto (character).
# set vars
x <- 1
y <- 'a'
# check classes
class(x)R> [1] "numeric"
class(y)R> [1] "character"Outra maneira de conhecer melhor um objeto é verificar a sua representação em texto. Todo objeto no R possui uma representação textual e a verificação desta é realizada através da função str:
R> int [1:10] 1 2 3 4 5 6 7 8 9 10
R> NULLEssa função é particularmente útil quando se está tentando entender os detalhes de um objeto mais complexo, tal como uma tabela. A utilidade da representação textual é que nela aparece o tamanho do objeto e suas classes internas. Nesse caso, o objeto x é da classe integer e possui dez elementos.
2.12 Mostrando e Formatando Informações na Tela
Como já vimos, é possível mostrar o valor de uma variável na tela de duas formas, digitando o nome dela no prompt ou então utilizando a função print. Explicando melhor, a função print é voltada para a apresentação de objetos e pode ser customizada para qualquer tipo. Por exemplo, caso tivéssemos um objeto de classe chamada MyTable que representasse um objeto tabular, poderíamos criar uma função chamada print.MyTable que irá mostrar uma tabela na tela com um formato especial tal como número de linhas, nomes das colunas, etc. A função print, portanto, pode ser customizada para cada classe de objeto.
Porém, existem outras funções específicas para apresentar texto (e não objetos) no prompt. A principal delas é message. Essa toma como input um texto, processa-o para símbolos específicos e o apresenta na tela. Essa função é muito mais poderosa e personalizável do que print.
Por exemplo, caso quiséssemos mostrar na tela o texto 'O valor de x é igual a 2', poderíamos fazê-lo da seguinte forma:
# set var
x <- 2
# print with message()
message('The value of x is', x)R> The value of x is2Função message também funciona para vetores:
# set vec
x <- 2:5
# print with message()
message('The values in x are: ', x)R> The values in x are: 2345A customização da saída da tela é possível através de comandos específicos. Por exemplo, se quiséssemos quebrar a linha da tela, poderíamos fazê-lo através do uso do caractere reservado \n:
# set char
my_text <- 'First line,\nSecond Line,\nThird Line'
# print with new lines
message(my_text)R> First line,
R> Second Line,
R> Third LineObserve que o uso do print não resultaria no mesmo efeito, uma vez que esse comando apresenta o texto como ele é, sem processar para efeitos específicos:
print(my_text)R> [1] "First line,\nSecond Line,\nThird Line"Outro exemplo no uso de comandos específicos para texto é adicionar um espaçamento tab no texto apresentado com o símbolo \t. Veja a seguir:
# set char with \t
my_text_1 <- 'A and B'
my_text_2 <- '\tA and B'
my_text_3 <- '\t\tA and B'
# print with message()
message(my_text_1)R> A and B
message(my_text_2)R> A and B
message(my_text_3)R> A and BVale destacar que, na grande maioria dos casos de pesquisa, será necessário apenas o uso de \n para formatar textos de saída. Outras maneiras de manipular a saída de texto no prompt com base em símbolos específicos são encontradas no manual oficial do R.
Parte do processo de apresentação de texto na tela é a customização do mesmo. Para isto, existem duas funções muito úteis: paste e format.
A função paste cola uma série de caracteres juntos. É uma função muito útil, a qual será utilizada intensamente para o resto dos exemplos deste livro. Observe o código a seguir:
# set chars
my_text_1 <- 'I am a text'
my_text_2 <- 'very beautiful'
my_text_3 <- 'and informative.'
# using paste and message
message(paste(my_text_1, my_text_2, my_text_3))R> I am a text very beautiful and informative.O resultado anterior não está muito longe do que fizemos no exemplo com a função print. Note, porém, que a função paste adiciona um espaço entre cada texto. Caso não quiséssemos esse espaço, poderíamos usar a função paste0:
R> I am a textvery beautifuland informative.
Uma alternativa a função message é cat
(concatenate and print). Não é incomum encontrarmos códigos
onde mensagens para o usuário são transmitidas via cat e
não message. Como regra, dê preferência a
message pois esta é mais fácil de controlar. Por exemplo,
caso o usuário quiser silenciar uma função, omitindo todas saídas da
tela, bastaria usar o comando suppressMessages.
Outra possibilidade muito útil no uso do paste é modificar o texto entre a junção dos itens a serem colados. Por exemplo, caso quiséssemos adicionar uma vírgula e espaço (,) entre cada item, poderíamos fazer isso através do uso do argumento sep, como a seguir:
R> I am a text, very beautiful, and informative.Caso tivéssemos um vetor atômico com os elementos da frase em um objeto apenas, poderíamos atingir o mesmo resultado utilizando paste o argumento collapse:
# using paste with collapse argument
my_text <-c('Eu sou um texto', 'muito bonito', 'e charmoso.')
message(paste(my_text, collapse = ', '))R> Eu sou um texto, muito bonito, e charmoso.Prosseguindo, o comando format é utilizado para formatar números e datas. É especialmente útil quando formos montar tabelas e buscarmos apresentar os números de uma maneira visualmente atraente. Por definição, o R apresenta uma série de dígitos após a vírgula:
# message without formatting
message(1/3)R> 0.333333333333333Caso quiséssemos apenas dois dígitos aparecendo na tela, utilizaríamos o seguinte código:
R> 0.33Tal como, também é possível mudar o símbolo de decimal:
R> 0,3333333Tal flexibilidade é muito útil quando devemos reportar resultados respeitando algum formato local tal como o Brasileiro.
Uma alternativa recente e muito interessante para o comando base::paste é stringr::str_c e stringr::str_glue. Enquanto a primeira é quase idêntica a paste0, a segunda tem uma maneira pecular de juntar objetos. Veja um exemplo a seguir:
library(stringr)
# define some vars
my_name <- 'Pedro'
my_age <- 23
# using base::paste0
my_str_1 <- paste0('My name is ', my_name, ' and my age is ', my_age)
# using stringr::str_c
my_str_2 <- str_c('My name is ', my_name, ' and my age is ', my_age)
# using stringr::str_glue
my_str_3 <- str_glue('My name is {my_name} and my age is {my_age}')
identical(my_str_1, my_str_2)R> [1] TRUE
identical(my_str_1, my_str_3)R> [1] FALSE
identical(my_str_2, my_str_3)R> [1] FALSEComo vemos, temos três alternativas para o mesmo resultado final. Note que str_glue usa de chaves para definir as variáveis dentro do próprio texto. Esse é um formato muito interessante e prático.
2.13 Conhecendo o Tamanho dos Objetos
Na prática de programação com o R, é muito importante saber o tamanho das variáveis que estão sendo utilizadas. Isso serve não somente para auxiliar o usuário na verificação de possíveis erros do código, mas também para saber o tamanho necessário em certos procedimentos de iteração tal como loops, os quais serão tratados em capítulo futuro.
No R, o tamanho do objeto pode ser verificado com o uso de quatro principais funções: length, nrow, ncol e dim.
A função length é destinada a objetos com uma única dimensão, tal como vetores atômicos:
# set x
x <- c(2,3,3,4,2,1)
# get length x
n <- length(x)
# display message
message(paste('The length of x is', n))R> The length of x is 6Para objetos com mais de uma dimensão, por exemplo matrizes e dataframes, utilizam-se as funções nrow, ncol e dim para descobrir o número de linhas (primeira dimensão) e o número de colunas (segunda dimensão). Veja a diferença a seguir.
R> [,1] [,2] [,3] [,4] [,5]
R> [1,] 1 5 9 13 17
R> [2,] 2 6 10 14 18
R> [3,] 3 7 11 15 19
R> [4,] 4 8 12 16 20
# find number of rows, columns and elements
my_nrow <- nrow(x)
my_ncol <- ncol(x)
my_length <- length(x)
# print message
message(paste('\nThe number of lines in x is ', my_nrow))R>
R> The number of lines in x is 4R>
R> The number of columns in x is 5R>
R> The number of elements in x is 20Já a função dim mostra a dimensão do objeto, resultando em um vetor numérico como saída. Essa deve ser utilizada quando o objeto tiver mais de duas dimensões. Na prática, esses casos são raros. Um exemplo para a variável x é dado a seguir:
R> [1] 4 5Para o caso de objetos com mais de duas dimensões, podemos utilizar a função array para criá-los e dim para descobrir o seu tamanho:
R> , , 1
R>
R> [,1] [,2] [,3]
R> [1,] 1 4 7
R> [2,] 2 5 8
R> [3,] 3 6 9
R>
R> , , 2
R>
R> [,1] [,2] [,3]
R> [1,] 1 4 7
R> [2,] 2 5 8
R> [3,] 3 6 9
R>
R> , , 3
R>
R> [,1] [,2] [,3]
R> [1,] 1 4 7
R> [2,] 2 5 8
R> [3,] 3 6 9R> [1] 3 3 3Uma observação importante aqui é que as funções length, nrow, ncol e dim não servem para descobrir o número de letras em um texto. Esse é um erro bastante comum. Por exemplo, caso tivéssemos um objeto do tipo texto e usássemos a função length, o resultado seria o seguinte:
# set char object
my_char <- 'abcde'
# find its length (and NOT number of characters)
print(length(my_char))R> [1] 1Isso ocorre pois a função length retorna o número de elementos. Nesse caso, my_char possui apenas um elemento. Para descobrir o número de caracteres no objeto, utilizamos a função nchar, conforme a seguir:
R> [1] 5Reforçando, cada objeto no R tem suas propriedades e funções específicas para manipulação.
2.14 Selecionando Elementos de um Vetor Atômico
Após a criação de um vetor atômico de qualquer classe, é possível que se esteja interessado em apenas um ou alguns elementos desse mesmo vetor. Por exemplo, caso estivéssemos buscando atualizar o valor de um portfólio de investimento, o nosso interesse dentro de um vetor contendo preços de uma ação é somente para o preço mais recente. Todos os demais preços não seriam relevantes para a nossa análise e, portanto, poderiam ser ignorados.
Esse processo de seleção de pedaços de um vetor atômico é chamado de indexação e é executado através do uso de colchetes []. Observe o exemplo de código a seguir:
# set my_x
my_x <- c(1, 5, 4, 3, 2, 7, 3.5, 4.3)Se quiséssemos apenas o terceiro elemento de my_x, utilizaríamos o operador de colchete da seguinte forma:
# get third element of my_x
elem_x <- my_x[3]
print(elem_x)R> [1] 4Também podemos utilizar o comando length, apresentado anteriormente, para acessar o último elemento do vetor:
R> [1] 4.3No caso de estarmos interessado apenas no último e penúltimo valor de my_x utilizaríamos o operador de sequência (:):
# get last and second last elements
piece_x_1 <- my_x[ (length(my_x)-1):length(my_x) ]
# print it
print(piece_x_1)R> [1] 3.5 4.3Uma propriedade única da linguagem R é que, caso for acessado uma posição que não existe no vetor, o programa retorna o valor NA (not available). Veja a seguir, onde tenta-se obter o quarto valor de um vetor com apenas três elementos.
R> [1] NAÉ importante conhecer esse comportamento do R, pois o não tratamento desses erros pode gerar problemas difíceis de identificar em um código mais complexo. Em outras linguagens de programação, a tentativa de acesso a elementos não existentes geralmente retorna um erro e cancela a execução do resto do código. No caso do R, dado que o acesso a elementos inexistentes não gera erro, é possível que isso gere um problema em outras partes do script.
Geralmente, a ocorrência de NAs (Not Available)
sugere a existência de problema no código. Saiba que NA
indicam a falta de dados e são contagiosos: tudo que interagir com
objeto do tipo NA, seja uma soma ou multiplicação, irá
também virar NA. O usuário deve prestar atenção
toda vez que surgirem valores NA de forma inesperada nos
objetos criados. Uma inspeção nos índices dos vetores pode ser
necessária.
O uso de indexadores é muito útil quando se está procurando por itens de um vetor que satisfaçam alguma condição. Por exemplo, caso quiséssemos todos os valores de my_x que são maiores que 3, utilizaríamos o seguinte comando:
# get all values higher than 3
piece_x_2 <- my_x[my_x>3]
# print it
print(piece_x_2)R> [1] 5.0 4.0 7.0 3.5 4.3É possível também indexar por mais de uma condição através dos operadores de lógica & (e) e | (ou). Por exemplo, caso quiséssemos os valores de my_x maiores que 2 e menores que 4, usaríamos o seguinte comando:
# get all values higher than 2 AND lower than 4
piece_x_3 <- my_x[ (my_x>2) & (my_x<4) ]
# print it
print(piece_x_3)R> [1] 3.0 3.5Da mesma forma, havendo interesse nos itens que são menores que 3 ou maiores que 6, teríamos:
# get all values lower than 3 OR higher than 6
piece_x_4 <- my_x[ (my_x<3)|(my_x>6) ]
# print it
print(piece_x_4)R> [1] 1 2 7A indexação lógica também funciona com a interação de diferentes variáveis, isto é, podemos utilizar uma condição lógica em uma variável para selecionar itens em outra:
# set my_x and my_y
my_x <- c(1, 4, 6, 8, 12)
my_y <- c(-2, -3, 4, 10, 14)
# find elements in my_x where my_y are positive
my_piece_x <- my_x[ my_y > 0 ]
# print it
print(my_piece_x)R> [1] 6 8 12Olhando mais de perto o processo de indexação, vale salientar que, quando utilizamos uma condição de indexação dos dados, esta-se criando uma variável do tipo lógica. Essa toma apenas dois valores: TRUE (verdadeiro) ou FALSE (falso). É fácil perceber isso quando criamos o teste lógico em um objeto e o mostramos na tela:
# set logical object
my_logical <- my_y > 0
# print it
print(my_logical)R> [1] FALSE FALSE TRUE TRUE TRUE
# show its class
class(my_logical)R> [1] "logical"As demais propriedades e operações com vetores lógicos serão explicadas em capítulo futuro.
2.15 Limpando a Memória
Após a criação de diversas variáveis, o ambiente de trabalho do R pode ficar cheio de conteúdo já utilizado e dispensável. Nesse caso, é desejável limpar a memória do programa. Geralmente isso é realizado no começo de um script, de forma que toda vez que o script for executado, a memória estará totalmente limpa antes de qualquer cálculo. Além de desocupar a memória do computador, isso ajuda a evitar possíveis erros no código. Na grande maioria dos casos, porém, a limpeza do ambiente de trabalho deve ser realizada apenas uma vez.
Por exemplo, dada uma variável x, podemos excluí-la da memória com o comando rm, conforme mostrado a seguir:
# set x and y
x <- 1
y <- 2
# print all existing objects
ls()
# remove x from memory
rm('x')
# print objects again
ls()Observe que o objeto x não estará mais mais disponível após o uso do comando rm('x').
Entretanto, em situações práticas é desejável limpar toda a memória utilizada por todos os objetos disponíveis no R. Pode-se atingir esse objetivo com o seguinte código:
O termo list é um argumento da função rm, o qual define quais objetos serão eliminados. Já o comando ls() mostra todas os objetos disponíveis atualmente. Portanto, ao encadear ambos os comandos, limpamos da memória todos os objetos disponíveis para o R. Como comentado, uma boa política de programação é sempre iniciar o script limpando a memória do R. .
A limpeza da memória em scripts é uma estratégia controversa. Alguns autores argumentam que é melhor não limpar a memória pois isso pode apagar resultados importantes. Na minha opinião, acho fundamental limpar a memória, desde que todos resultados sejam reproduzíveis. Ao iniciar um código sempre do mesmo estado, isto é, nenhuma variável criada, fica mais fácil de entender e capturar possíveis bugs.
2.16 Mostrando e Mudando o Diretório de Trabalho
Assim como outros softwares, o R sempre trabalha em algum diretório. É com base nesse diretório que o R procura arquivos para importar dados. É nesse mesmo diretório que o R salva arquivos, caso não definirmos um endereço no computador explicitamente. Essa saída pode ser um arquivo de uma figura, um arquivo de texto ou uma planilha eletrônica. Como boa prática de criação e organização de scripts, deve-se sempre mudar o diretório de trabalho para onde o arquivo do script está localizado.
Em sua inicialização, o R possui como diretório default a pasta de documentos do usuário cujo atalho é o tilda ('~').
Para mostrar o diretório atual de trabalho, basta utilizar a função getwd:
R> [1] home/msperlin/adfeR/01-Book ContentO resultado do código anterior mostra a pasta onde este livro foi escrito. Esse é o diretório onde os arquivos do livro foram compilados dentro do ambiente Linux.
A mudança de diretório de trabalho é realizada através do comando setwd. Por exemplo, caso quiséssemos mudar o nosso diretório de trabalho para C:/Minha pesquisa/, basta digitar no prompt:
# set dir
my_d <- 'C:/Minha Pesquisa/'
setwd(my_d)Enquanto para casos simples, como o anterior, lembrar o nome do diretório é fácil, em casos práticos o diretório de trabalho pode ser em um lugar mais profundo da raiz de diretórios do sistema de arquivos. Nessa situação, uma estratégia eficiente para descobrir a pasta de trabalho é utilizar um explorador de arquivos, tal como o Explorer no Windows. Abra esse aplicativo e vá até o local onde quer trabalhar com o seu script. Após isso, coloque o cursor na barra de endereço e selecione todo o endereço. Aperte control+c para copiar o endereço para a área de transferência. Volte para o seu código e cole o mesmo no código. Atenção nesta etapa, o Windows utiliza a barra invertida para definir endereços no computador, enquanto o R utiliza a barra normal. Caso tente utilizar a barra invertida, um erro será mostrado na tela. Veja o exemplo a seguir.
my_d <- 'C:\Minha pesquisa\'
setwd(my_d)O erro terá a seguinte mensagem:
Error: '\M' is an unrecognized escape in character string..."A justificativa para o erro é que a barra invertida \ é um caractere reservado no R e não pode ser utilizado isoladamente. Caso precises, podes definí-lo no objeto de texto com dupla barra, tal como em \\. Veja no exemplo a seguir, onde a dupla barra é substituída por uma barra única:
# set char with \
my_char <- 'using \\'
# print it
message(my_char)R> using \A solução do problema é simples. Após copiar o endereço, modifique todas as barras para a barra normal, assim como no código a seguir:
my_d <- 'C:/Minha pesquisa/'
setwd(my_d)É possível também utilizar barras invertidas duplas \\ na definição de diretórios, porém não se recomenda essa formatação, pois não é compatível com outros sistemas operacionais.
Outro ponto importante aqui é o uso de endereços relativos. Por exemplo, caso esteja trabalhando em um diretório que contém um subdiretório chamado Data, podes entrar nele com o seguinte código:
# change to subfolder
setwd('Data')Outra possibilidade pouco conhecida no uso de setwd é que é possível entrar em níveis inferiores do sistema de diretórios com .., tal como em:
# change to previous level
setwd('..')Portanto, caso estejas trabalhando no diretório C:/My Research/ e executar o comando setwd('..'), o diretório atual de trabalho viraria C:/, um nível inferior a C:/My Research/.
Uma maneira mais moderna e pouco conhecida de definir o diretório de trabalho é usar as funções internas do RStudio. Este é um conjunto de funções que só funcionam dentro do RStudio e fornecem diversas informações sobre o arquivo sendo editado. Para descobrir o caminho do arquivo atual que está sendo editado no RStudio e configurar o diretório de trabalho para lá, você pode escrever:
my_path <- dirname(rstudioapi::getActiveDocumentContext()$path)
setwd(my_path)Dessa forma, o script mudará o diretório para sua própria localização. Apesar de não ser um código exatamente elegante, ele é bastante funcional. Caso copie o arquivo para outro diretório, o valor de my_path muda para o novo diretório. Esteja ciente, no entanto, de que esse truque só funciona no editor de rotinas do RStudio e dentro de um arquivo salvo. O código não funcionará a partir do prompt.
Outro truque bastante útil para definir diretórios de trabalho no R é
usar o símbolo ~. Esse define a pasta
‘Documentos’ no Windows, a qual é única para cada
usuário. Portanto, ao executar setwd(‘~’), irás direcionar
o R a uma pasta de fácil acesso e livre modificação pelo usuário atual
do computador.
2.17 Comentários no Código
Comentários são definidos usando o símbolo #. Qualquer texto a direita desse símbolo não será processado pelo R. Note que até a cor do código a direita do hashtag muda no RStudio. Isso dá liberdade para escrever o que for necessário dentro do script. Um exemplo:
# This is a comment
# This is another comment
x <- 'abc' # this is another comment, but mixed with code
my_l <- list(var1 = 1:10, # set var 1
var2 = 2:5) # another varOs comentários são uma eficiente maneira de comunicar qualquer informação importante que não pode ser inferida diretamente do código. O uso correto de comentários é tão importante quanto o código em si. Quando bem feitos, aumentam a reproducibilidade, organização e entendimento do código. Em geral, você deve evitar o uso de comentários que são muito óbvios ou muito genéricos. Por exemplo:
# read a csv file
df <- read.csv ('MyDataFile.csv')Como você pode ver, é bastante óbvio que a linha df <- read.csv('MyDataFile.csv') está lendo um arquivo .csv. O nome da função, read.csv já afirma isso. Então, o comentário não foi bom pois não adicionou novas informações ao usuário. Uma melhor abordagem seria definir o autor, a descrição da funcionalidade do script e explicar melhor a origem e a última atualização do arquivo de dados. Vamos dar uma olhada:
# Script for reproducing results of JOHN (2018)
# Author: Mr Researcher (dontspamme@emailprovider.com)
# Last script update: 2018-01-10
#
# File downloaded from www.sitewithdatafiles.com/data-files/
# The description of the data goes here
#
# Last file update: 2017-12-05
df <- read.csv('MyDataFile.csv')Com esses comentários, o usuário saberá o propósito do script, quem o escreveu e a data da última edição. A origem do arquivo e a data de atualização mais recente também estão disponíveis. Se o usuário quiser atualizar os dados, tudo o que ele tem a fazer é ir ao mencionado site e baixar o novo arquivo. Isso facilitará o uso futuro e o compartilhamento do script.
Outro uso de comentários é definir seções no código, como em:
# Script for reproducing results of JOHN (2018)
# Author: Mr Researcher (dontspamme@emailprovider.com)
# Last script update: 2018-01-10
#
# File downloaded from www.sitewithdatafiles.com/data-files/
# The description of the data goes here
#
# Last file update: 2017-12-05
# Clean data -------------------------
# - remove outliers
# - remove unnecessary columns
# Create descriptive tables ----------
# Estimate models --------------------
# Report results ---------------------O uso de uma longa linha de traços (-) é intencional. Isto faz com que o RStudio identifique as seções do código e apresente no espaço abaixo do editor de rotinas um atalho para acessar as correspondentes linhas de cada seção. Teste você mesmo, copie e cole o código acima em um novo script do RStudio, salve o mesmo, e verás que as seções aparecem em um botão entre o editor e o prompt. Desta forma, uma vez que você precisa mudar uma parte específica do código, você pode se dirigir rapidamente a seção desejada.
Quando começar a compartilhar código com outras pessoas, logo perceberás que os comentários são essenciais e esperados. Eles ajudam a transmitir informações que não estão disponíveis no código. Uma nota aqui, ao longo do livro você verá que os comentários do código são, na maior parte do tempo, bastante óbvios. Isso foi intencional, pois mensagens claras e diretas são importantes para novos usuários, os quais fazem parte da audiência.
2.18 Cancelando a Execução de um Código
Toda vez que o R estiver executando algum código, uma sinalização visual no formato de um pequeno círculo vermelho no canto direito do prompt irá aparecer. Caso conseguir ler (o símbolo é pequeno em monitores modernos), o texto indica o termo stop. Esse símbolo não somente indica que o programa ainda está rodando mas também pode ser utilizado para cancelar a execução de um código. Para isso, basta clicar no referido botão. Outra maneira de cancelar uma execução é apontar o mouse no prompt e pressionar a tecla Esc no teclado.
Para testar o cancelamento de código, copie e cole o código a seguir em um script do RStudio. Após salvar, rode o mesmo com control+shift+s.
O código anterior usa um comando especial do tipo for para mostrar a mensagem a cada segundo. Neste caso, o código demorará 100 segundos para rodar. Caso não desejes esperar, aperte esc para cancelar a execução. Por enquanto, não se preocupe com as funções utilizadas no exemplo. Iremos discutir o uso do comando for no capítulo 8.
2.19 Procurando Ajuda
Uma tarefa muito comum no uso do R é procurar ajuda. A quantidade de funções disponíveis para o R é gigantesca e memorizar todas peculariedades é quase impossível. Assim, até mesmo usuários avançados comumente procuram ajuda sobre tarefas específicas no programa, seja para entender detalhes sobre algumas funções ou estudar um novo procedimento. Portanto, saibas que o uso do sistema de ajuda do R faz parte do cotidiano.
É possível buscar ajuda utilizando tanto o painel de help do RStudio como diretamente do prompt. Para isso, basta digitar o ponto de interrogação junto ao objeto sobre o qual se deseja ajuda, tal como em ?mean. Nesse caso, o objeto mean é uma função e o uso do comando irá abrir o painel de ajuda sobre ela.
No R, toda tela de ajuda de uma função é igual, conforme se vê na Figura 2.7 apresentada a seguir. Esta mostra uma descrição da função mean, seus argumentos de entrada explicados e também o seu objeto de saída. A tela de ajuda segue com referências e sugestões para outras funções relacionadas. Mais importante, os exemplos de uso da função aparecem por último e podem ser copiados e colados para acelerar o aprendizado no uso da função.

Figura 2.7: Tela de ajuda da função mean
Caso quiséssemos procurar um termo nos arquivos de ajuda, bastaria utilizar o comando ??"standard deviation". Essa operação irá procurar a ocorrência do termo em todos os pacotes do R e é muito útil para aprender como realizar alguma operação, nesse caso o cálculo de desvio padrão.
Como sugestão, o ponto inicial e mais direto para aprender uma nova função é observando o seu exemplo de uso, localizada no final da página de ajuda. Com isto, podes verificar quais tipos de objetos de entrada a mesma aceita e qual o formato e o tipo de objeto na sua saída. Após isso, leia atentamente a tela de ajuda para entender se a mesma faz exatamente o que esperas e quais são as suas opções de uso nas respectivas entradas. Caso a função realizar o procedimento desejado, podes copiar e colar o exemplo de uso para o teu próprio script, ajustando onde for necessário.
Outra fonte muito importante de ajuda é a própria internet. Sites como stackoverflow.com e mailing lists específicos do R, cujo conteúdo também está na internet, são fontes preciosas de informação. Havendo alguma dúvida que não foi possível solucionar via leitura dos arquivos de ajuda do R, vale o esforço de procurar uma solução via mecanismo de busca na internet. Em muitas situações, o seu problema, por mais específico que seja, já ocorreu e já foi solucionado por outros usuários.
Caso estiver recebendo uma mensagem de erro enigmática, outra dica é copiar e colar a mesma para uma pesquisa no Google. Aqui apresenta-se outro benefício do uso da língua inglesa. É mais provável que encontres a solução se o erro for escrito em inglês, dado o maior número de usuários na comunidade global. Caso não encontrar uma solução desta forma, podes inserir uma pergunta no stackoverflow ou no grupo Brasileiro do R no Facebook.
Toda vez que for pedir ajuda na internet, procure sempre 1) descrever claramente o seu problema e 2) adicionar um código reproduzível do seu problema. Assim, o leitor pode facilmente verificar o que está acontecendo ao rodar o exemplo no seu computador. Não tenho dúvida que, se respeitar ambas regras, logo uma pessoa caridosa lhe ajudará com o seu problema.
2.20 Utilizando Code Completion com a Tecla tab
Um dos recursos mais úteis do RStudio é o preenchimento automático de código (code completion). Essa é uma ferramenta de edição que facilita o encontro de nomes de objetos, nome de pacotes, nome de arquivos e nomes de entradas em funções. O seu uso é muito simples. Após digitar um texto qualquer, basta apertar a tecla tab e uma série de opções aparecerá. Veja a Figura 2.8 apresentada a seguir, em que, após digitar a letra f e apertar tab, aparece uma janela com uma lista de objetos que iniciam com a respectiva letra.
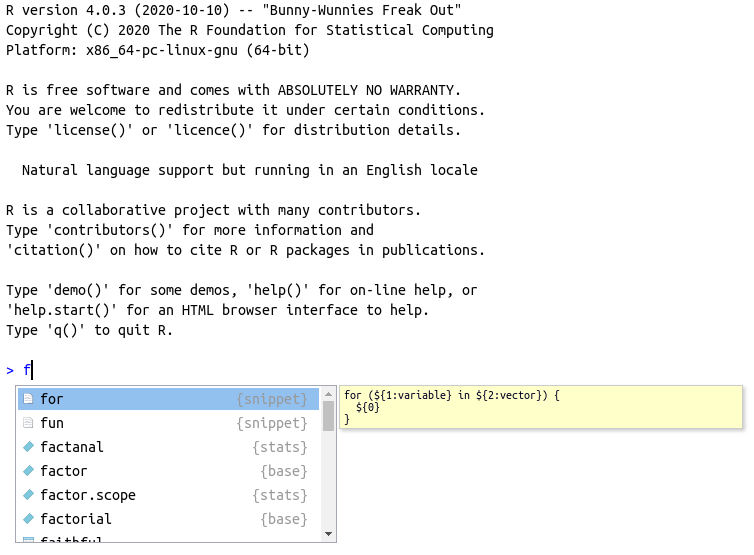
Figura 2.8: Uso do autocomplete para objetos
Essa ferramenta também funciona para pacotes. Para verificar, digite library(r) no prompt ou no editor, coloque o cursor entre os parênteses e aperte tab. O resultado deve ser algo parecido com a figura 2.9.
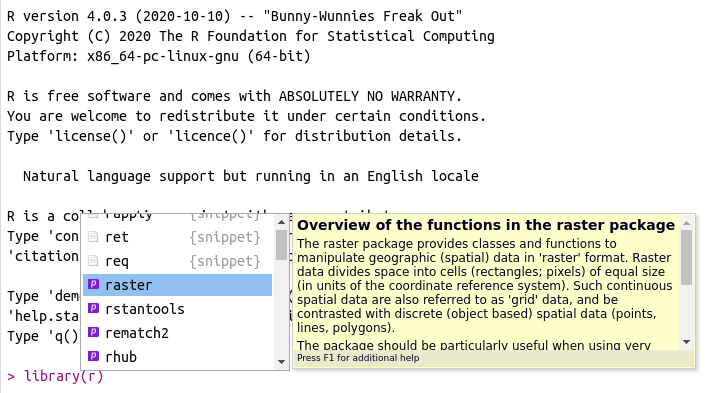
Figura 2.9: Uso do autocomplete para pacotes
Observe que uma descrição do pacote ou objeto também é oferecida. Isso facilita bastante o dia a dia, pois a memorização das funcionalidades e dos nomes dos pacotes e os objetos do R não é uma tarefa fácil. O uso do tab diminui o tempo de investigação dos nomes e evita possíveis erros de digitação na definição destes.
O uso dessa ferramenta torna-se ainda mais benéfico quando os objetos são nomeados com algum tipo de padrão. No restante do livro observarás que os objetos tendem a ser nomeados com o prefixo my, como em my_x, my_num. O uso desse padrão facilita o encontro futuro do nome dos objetos, pois basta digitar my, apertar tab e uma lista de todos os objetos criados pelo usuário aparecerá.
Outro uso do tab é no encontro de arquivos e pastas no computador. Basta criar uma variável como my_file <- " ", apontar o cursor para o meio das aspas e apertar a tecla tab. Uma tela com os arquivos e pastas do diretório atual de trabalho aparecerá, conforme mostrado na figura 2.10. Nesse caso específico, o R estava direcionado para a minha pasta de códigos, em que é possível enxergar diversos trabalhos realizados no passado.
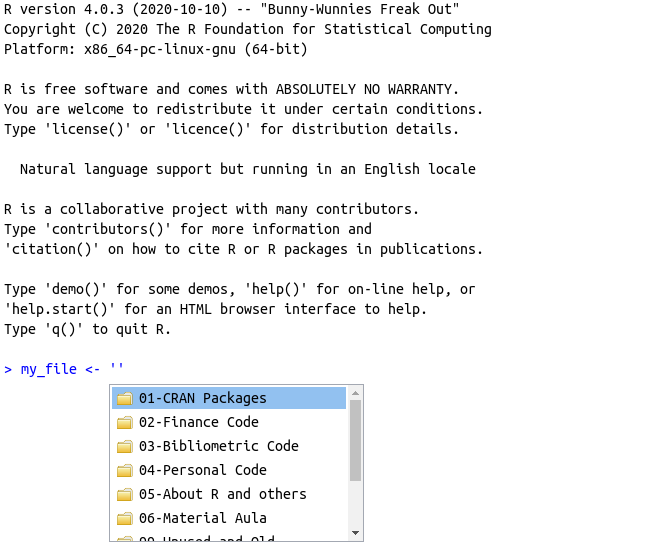
Figura 2.10: Uso do autocomplete para arquivos
Uma dica aqui é utilizar o tab com a raiz do computador. Assumindo que o disco do seu computador está alocado para C:/, digite my_file <- "C:/" e pressione tab após o símbolo /. Uma tela com os arquivos da raiz do computador aparecerá no RStudio. Podes facilmente navegar o sistema de arquivos utilizando as setas e enter.
O autocomplete também funciona para encontrar e definir as entradas de uma função. Por se tratar de um tópico mais avançado, deixamos o seu uso e demonstração para o capítulo 8.
O autocomplete é uma das ferramentas mais importantes do RStudio, funcionando para encontro de objetos, locais no disco rígido, pacotes e funções. Acostume-se a utilizar a tecla tab o quanto antes e logo verá como fica mais fácil escrever código rapidamente, e sem erros de digitação.
2.21 Interagindo com Arquivos e o Sistema Operacional
Em muitas situações de uso do R será necessário interagir com os arquivos do computador, seja criando novas pastas, descompactando e compactando arquivos, listando e removendo arquivos do disco rígido do computador ou qualquer outro tipo de operação. Na grande maioria dos casos, o interesse é na manipulação de arquivos contendo dados.
2.21.1 Listando Arquivos e Pastas
Para listar arquivos do computador, basta utilizar o função list.files. O argumento path define o diretório para listar os arquivos. Na construção deste livro foi criado um diretório chamado 00-text-resources/data, onde alguns dados são salvos. Pode-se verificar os arquivos nessa pasta com o seguinte código:
my_f <- list.files(path = "00-text-resources/data", full.names = TRUE)
print(my_f[1:5])R> [1] "00-text-resources/data/AdjustedPrices-InternacionalIndices.RDATA"
R> [2] "00-text-resources/data/BovStocks_2011-12-01_2016-11-29.csv"
R> [3] "00-text-resources/data/BovStocks_2011-12-01_2016-11-29.RData"
R> [4] "00-text-resources/data/example_gethfdata.RDATA"
R> [5] "00-text-resources/data/FileWithLatinChar_ISO-8859-9.txt"Observe que nesse diretório encontram-se vários arquivos .csv, .rds e .xlsx. Esses contêm dados que serão utilizados em capítulos futuros. Recomenda-se utilizar o argumento full.names como TRUE, o qual faz com que o retorno da função list.files contenha o caminho completo do arquivo. Isso facilita, por exemplo, uma possível importação de dados em que é necessário indicar não somente o nome do arquivo, mas a sua localização completa no computador. Destaca-se que também é possível listar os arquivos de forma recursiva, isto é, listar os arquivos de subpastas do endereço original. Para verificar, tente utilizar o seguinte código no seu computador:
# list all files recursively
list.files(path = getwd(), recursive = T, full.names = TRUE)O comando anterior irá listar todos os arquivos existentes na pasta atual e subpastas de trabalho. Dependendo de onde o comando foi executado, pode levar um certo tempo para o término do processo. Caso precisar cancelar a execução, aperte esc no teclado.
Para listar pastas (diretórios) do computador, basta utilizar o comando list.dirs. Veja a seguir.
R> [1] "./_book"
R> [2] "./_bookdown_files"
R> [3] "./.Rproj.user"
R> [4] "./00-code-resources"
R> [5] "./00-text-resources"
R> [6] "./adfeR_pt_ed03_cache"
R> [7] "./adfeR_pt_ed03_files"
R> [8] "./adfeR_pt_ed03-ONLINE_cache"
R> [9] "./adfeR_pt_ed03-ONLINE_files"
R> [10] "./gdfpd2_cache"
R> [11] "./gfred_cache"
R> [12] "./many_datafiles"
R> [13] "./quandl_cache"
R> [14] "./tabs"
R> [15] "./TD Files"No caso anterior, o comando lista todos os diretórios do trabalho atual sem recursividade. A saída do comando mostra os diretórios que utilizei para escrever este livro. Isso inclui o diretório de saída do livro (./_book), entre diversos outros. Nesse mesmo diretório, encontram-se os capítulos do livro, organizados por arquivos e baseados na linguagem RMarkdown (.Rmd). Para listar somente os arquivos com extensão .Rmd, utiliza-se o argumento pattern da função list.files, como a seguir:
list.files(path = getwd(), pattern = "*.Rmd$")R> [1] "_BemVindo.Rmd"
R> [2] "00a-Sobre-NovaEdicao.Rmd"
R> [3] "00b-Prefacio.Rmd"
R> [4] "01-Introducao.Rmd"
R> [5] "02-Operacoes-Basicas.Rmd"
R> [6] "03-Scripts-Pesquisa.Rmd"
R> [7] "04-Importacao-Exportacao-Local.Rmd"
R> [8] "05-Importacao-Internet.Rmd"
R> [9] "06-Objetos-Armazenamento.Rmd"
R> [10] "07-Objetos-Basicos.Rmd"
R> [11] "08-Programacao-com-R--ONLINE.Rmd"
R> [12] "09-Limpando-Estruturando-Dados--ONLINE.Rmd"
R> [13] "10-Figuras--ONLINE.Rmd"
R> [14] "11-Modelagem--ONLINE.Rmd"
R> [15] "12-Reportando-resultados--ONLINE.Rmd"
R> [16] "13-Otimizacao-código--ONLINE.Rmd"
R> [17] "14-Referencias.Rmd"
R> [18] "adfeR_pt_ed03-ONLINE.Rmd"
R> [19] "index.Rmd"O texto *.Rmd$ orienta o R a procurar todos arquivos que terminam o seu nome com o texto .Rmd. Os símbolos '*'' e '$' são operadores específicos para o encontro de padrões em texto em uma linguagem chamada regex (regular expressions) e, nesse caso, indicam que o usuário quer encontrar todos arquivos com extensão .Rmd. O símbolo ’*’ diz para ignorar qualquer texto anterior a ‘.Rmd’ e ‘$’ indica o fim do nome do arquivo. Os arquivos apresentados anteriormente contêm todo o conteúdo deste livro, incluindo este próprio parágrafo, localizado no arquivo 02-OperacoesBasicas.Rmd!
2.21.2 Apagando Arquivos e Diretórios
A remoção de arquivos é realizada através do comando file.remove:
# create temporary file
my_file <- 'MyTemp.csv'
write.csv(x = data.frame(x=1:10),
file = my_file)
# delete it
file.remove(my_file)R> [1] TRUELembre-se que deves ter permissão do seu sistema operacional para apagar um arquivo. Para o nosso caso, o retorno TRUE mostra que a operação teve sucesso.
Para deletar diretórios e todos os seus elementos, utilizamos unlink:
# create temp dir
dir.create('temp')
# fill it with file
my_file <- 'temp/tempfile.csv'
write.csv(x = data.frame(x=1:10),
file = my_file)
unlink(x = 'temp', recursive = TRUE)A função, neste caso, não retorna nada. Podes checar se o diretório existe com dir.exists:
dir.exists('temp')R> [1] FALSE
Não preciso nem dizer, tenha muito cuidado com
comandos file.remove e unlink, principalmente
quando utilizar a recursividade (recursive = TRUE). Uma
execução errada e partes importantes do seu disco rídigo podem ser
apagadas, deixando o seu computador inoperável. Vale salientar que o R
realmente apaga os arquivos e não somente manda para a
lixeira. Portanto, ao apagar diretórios com unlink, não
poderás recuperar os arquivos.
2.21.3 Utilizando Arquivos e Diretórios Temporários
Um aspecto interessante do R é que ele possui uma pasta temporária que é criado na inicialização do programa. Esse diretório serve para guardar quaisquer arquivos descartáveis gerados pelo R. A cada nova sessão do R, um novo diretório temporário é criado. Ao inicializarmos o computador, essa pasta temporária é deletada.
O endereço do diretório temporário de uma sessão do R é verificado com tempdir:
R> My tempdir is /tmp/RtmpN2n71FO último texto do diretório, neste caso RtmpN2n71F é aleatóriamente definido e irá trocar a cada nova sessão do R.
A mesma dinâmica é encontrada para nomes de arquivos. Caso queira, por algum motivo, utilizar um nome temporário e aleatório para algum arquivo com extensão .txt, utilize tempfile e defina a entrada fileext:
R> /tmp/RtmpN2n71F/file11cf43d05d89.txtNote que o nome do arquivo – file11cf43d05d89.txt – é totalmente aleatório e mudará a cada chamada de tempfile.
2.21.4 Baixando Arquivos da Internet
O R pode baixar arquivos da Internet diretamente no código. Isso é realizado com a função download.file. Veja o exemplo a seguir, onde baixamos uma planilha de Excel do site da Microsoft para um arquivo temporário:
# set link
link_dl <- 'go.microsoft.com/fwlink/?LinkID=521962'
local_file <- tempfile(fileext = '.xlsx') # name of local file
download.file(url = link_dl,
destfile = local_file)O uso de download.file é bastante prático quando se está trabalhando com dados da Internet que são constantemente atualizados. Basta baixar e atualizar o arquivo com dados no início do script. Poderíamos continuar a rotina lendo o arquivo baixado e realizando a nossa análise dos dados disponíveis.
Um exemplo nesse caso é a tabela de empresas listadas na bolsa divulgada pela CVM (comissão de valores mobiliários). A tabela está disponível em um arquivo no site. Podemos baixar o arquivo e, logo em seguida, ler os dados.
library(readr)
library(dplyr)
# set destination link and file
my_link <- 'http://dados.cvm.gov.br/dados/CIA_ABERTA/CAD/DADOS/cad_cia_aberta.csv'
my_destfile <- tempfile(fileext = '.csv')
# download file
download.file(my_link,
destfile = my_destfile,
mode = "wb")
# read it
df_cvm <- read_csv2(my_destfile,
#delim = '\t',
locale = locale(encoding = 'Latin1'),
col_types = cols())R> ℹ Using "','" as decimal and "'.'" as grouping mark. Use `read_delim()` for more control.R> [1] "CNPJ_CIA" "DENOM_SOCIAL"
R> [3] "DENOM_COMERC" "DT_REG"
R> [5] "DT_CONST" "DT_CANCEL"
R> [7] "MOTIVO_CANCEL" "SIT"
R> [9] "DT_INI_SIT" "CD_CVM"
R> [11] "SETOR_ATIV" "TP_MERC"
R> [13] "CATEG_REG" "DT_INI_CATEG"
R> [15] "SIT_EMISSOR" "DT_INI_SIT_EMISSOR"
R> [17] "CONTROLE_ACIONARIO" "TP_ENDER"
R> [19] "LOGRADOURO" "COMPL"
R> [21] "BAIRRO" "MUN"
R> [23] "UF" "PAIS"
R> [25] "CEP" "DDD_TEL"
R> [27] "TEL" "DDD_FAX"
R> [29] "FAX" "EMAIL"
R> [31] "TP_RESP" "RESP"
R> [33] "DT_INI_RESP" "LOGRADOURO_RESP"
R> [35] "COMPL_RESP" "BAIRRO_RESP"
R> [37] "MUN_RESP" "UF_RESP"
R> [39] "PAIS_RESP" "CEP_RESP"
R> [41] "DDD_TEL_RESP" "TEL_RESP"
R> [43] "DDD_FAX_RESP" "FAX_RESP"
R> [45] "EMAIL_RESP" "CNPJ_AUDITOR"
R> [47] "AUDITOR"Existem diversas informações interessantes nestes dados incluindo nome e CNPJ de empresas listadas (ou deslistadas) da bolsa de valores Brasileira. E, mais importante, o arquivo está sempre atualizado. O código anterior estará sempre buscando os dados mais recentes a cada execução.
2.22 Exercícios
Q.1
Crie um novo script, salve o mesmo em uma pasta pessoal. Agora, escreva os comandos no script que definam dois objetos: um contendo uma sequência entre 1 e 100 e outro com o texto do seu nome (ex. 'Ricardo'). Execute o código com os atalhos no teclado.
x <- 1:100
y <- 'Ricardo'
# press control+shift+enter to run this chunk of code in RStudio
Q.2
No script criado anteriormente, use função message para mostrar a seguinte frase no prompt do R: "My name is ....".
x <- 36
y <- 'Ricardo'
message(paste0('My name is ', y))
# press control+shift+enter to run this chunk of code in RStudio
Q.3
Dentro do mesmo script, mostre o diretório atual de trabalho (veja função getwd, tal como em print(getwd())). Agora, modifique o seu diretório de trabalho para o Desktop (Área de Trabalho) e mostre a seguinte mensagem na tela do prompt: 'My desktop address is ....'. Dica: use e abuse da ferramenta autocomplete do RStudio para rapidamente encontrar a pasta do desktop.
current_dir <- getwd()
print(current_dir)
new_dir <- '~/Desktop/' # this is probably C:/Users/USERNAME/Desktop for Windows
setwd(new_dir)
cat(paste0('My desktop address is ', getwd()))
Q.4
Utilize o R para baixar o arquivo compactado com o material do livro, disponível nesse link14. Salve o mesmo como um arquivo na pasta temporária da sessão (veja função tempfile).
local_file <- tempfile(fileext = '.zip')
my_url <- 'https://www.msperlin.com/files/pafdr%20files/Code_Data_pafdR.zip'
download.file(url = my_url,
destfile = local_file)
# check if exists
file.exists(local_file)
Q.5
Utilize a função unzip para descompactar o arquivo baixado na questão anterior para um diretório chamado 'adfeR-Files' dentro da pasta do “Desktop”. Quantos arquivos estão disponíveis na pasta resultante? Dica: use o argumento recursive = TRUE com list.files para procurar também todos subdiretórios disponíveis.
my_folder <- '~/Desktop/adfeR-Files' # this is probably C:/Users/USERNAME/Desktop for Windows
unzip(local_file, exdir = my_folder) # local_file comes from previous exercise
files <- list.files(my_folder,
full.names = TRUE,
recursive = TRUE)
n_files <- length(files)
message(paste0('There are ', n_files, ' files available at folder "', my_folder, '".'))
Q.6
Toda vez que o usuário instala um pacote do R, os arquivos particulares ao pacote são armazenados localmente em uma pasta específica do computador. Utilizando comando Sys.getenv('R_LIBS_USER') e list.dirs, liste todos os diretórios desta pasta. Quantos pacotes estão disponíveis nesta pasta do seu computador?
r_pkg_folder <- Sys.getenv ('R_LIBS_USER')
available_dirs <- list.dirs(r_pkg_folder, recursive = FALSE)
n_dirs <- length(available_dirs)
cat(paste0('There are ', n_dirs, ' folders available at "', r_pkg_folder, '".'))
Q.7
No mesmo assunto do exercício anterior, liste todos os arquivos em todas as subpastas do diretório contendo os arquivos dos diferentes pacotes. Em média, quantos arquivos são necessários para cada pacote?
r_pkg_folder <- Sys.getenv ('R_LIBS_USER')
pkg_files <- list.files(r_pkg_folder, recursive = TRUE)
my_dirs <- list.dirs(r_pkg_folder, recursive = FALSE)
n_files <- length(pkg_files)
n_dirs <- length(my_dirs)
my_msg <- paste0('We have ', length(pkg_files), ' ',
'files for ', length(my_dirs), ' packages. \n',
'On average, there are ', n_files/n_dirs, ' files per directory.')
message(my_msg)
Q.8
Use função install.packages para instalar o pacote BatchGetSymbols no seu computador. Após a instalação, use função BatchGetSymbols::BatchGetSymbols para baixar dados de preços para a ação da Petrobrás – PETR3 (PETR3.SA no Yahoo finance) – nos últimos 15 dias. Dicas: 1) use função Sys.Date() para definir data atual e Sys.Date() - 15 para calcular a data localizada 15 dias no passado; 2) note que a saída de BatchGetSymbols é uma lista, um tipo especial de objeto, e o que os dados de preços estão localizados no segundo elemento dessa lista.
if (!require(BatchGetSymbols)) install.packages('BatchGetSymbols')
l_out <- BatchGetSymbols(tickers = 'PETR3.SA',
first.date = Sys.Date() - 15,
last.date = Sys.Date())
df_prices <- l_out[[2]]
str(df_prices)
Q.9
O pacote cranlogs permite o acesso a estatísticas de downloads de pacotes do CRAN. Após instalar o cranlogs no seu computador, use função cranlogs::cran_top_downloads para verificar quais são os 10 pacotes mais instalados pela comunidade global no último mês. Qual o pacote em primeiro lugar? Dica: Defina a entrada da função cran_top_downloads como sendo when = 'last-month'. Também note que a resposta aqui pode não ser a mesma que obteve pois esta depende do dia em que foi executado o código.
Resposta:
#if (!require(cranlogs)) install.packages('cranlogs')
pkgs <- cranlogs::cran_top_downloads(when = 'last-month')
my_sol <- pkgs$package[1]
Q.10
Utilizando pacote devtools, instale a versão de desenvolvimento do pacote ggplot2, disponível no repositório de Hadley Hickman. Carregue o pacote usando library e crie uma figura simples com o código qplot(y = rnorm(10), x = 1:10).
if (!require(devtools)) install.packages("devtools")
devtools::install_github('hadley/ggplot2')
library(ggplot2)
qplot(y = rnorm (10), x = 1:10)
Q.11
Utilizando sua capacidade de programação, verifique no seu computador qual pasta, a partir do diretório de “Documentos” (atalho = ~), possui o maior número de arquivos. Apresente na tela do R as cinco pastas com maior número de arquivos.
doc_folder <- '~' # 'C:/Users/USERNAME/Documents' in Windows
# '/home/USERNAME/ in Linux
fct_count_files <- function(dir_in) {
n_files <- list.files(dir_in, recursive = FALSE)
return(length(n_files))
}
# be aware this might take lots of time...
all_folders <- fs::dir_ls(path = doc_folder,
type = 'directory',
recurse = TRUE)
counter_files <- sapply(all_folders, fct_count_files)
sorted <- sort(counter_files, decreasing = TRUE)
message('\nThe five folders with highest number of files are:\n\n')
message(paste0(names(sorted[1:5]), collapse = '\n'))